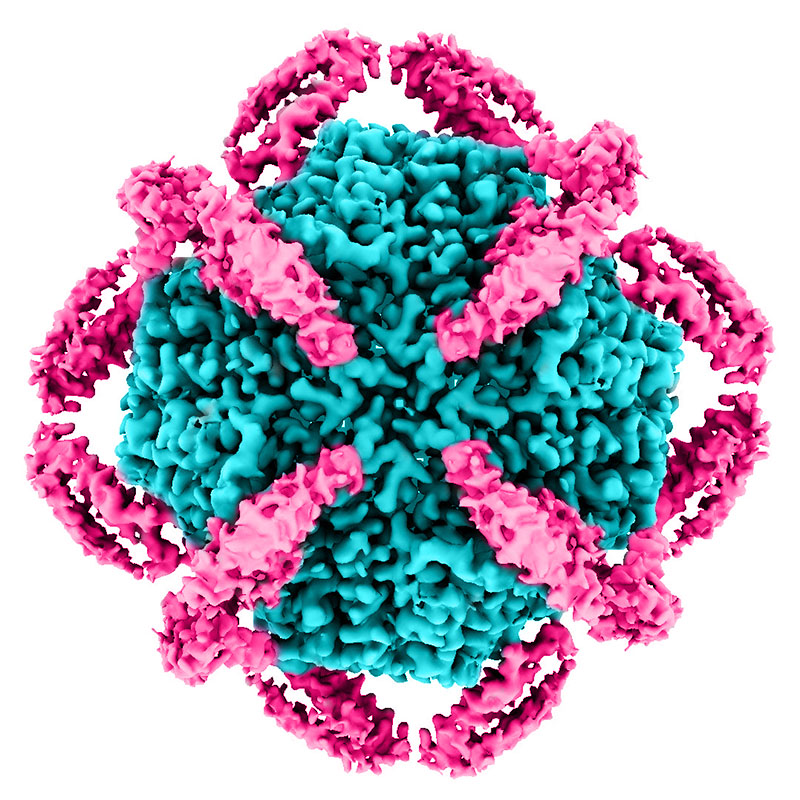
Reconstruction cryo-EM détaillée des protéines KIX (magenta) entourant la coquille centrale d’apoferritine (cyan). L’enveloppe externe de la MBP n’est pas représentée. Crédit : Greg Stewart/SLAC National Accelerator Laboratory.
La prise en sandwich de protéines ondulantes entre deux autres couches permet aux scientifiques d’obtenir les images les plus détaillées à ce jour d’une protéine qui joue un rôle clé dans la propagation de la leucémie myéloïde aiguë.
Selon l’American Cancer Institute, la leucémie myéloïde aiguë (LMA) touchera plus de 20 000 Américains cette année, tuant plus de 11 000 d’entre eux. De nombreuses personnes traitées par chimiothérapie intensive ou par greffe de cellules souches peuvent subir des effets secondaires tels que des infections, la perte de cheveux et des vomissements, ainsi que des complications à long terme.
Kathleen Sakamoto, professeur à l’école de médecine de Stanford, a travaillé à la mise au point de médicaments contre la LMA et d’autres maladies du sang pour tenter de remédier à cette situation. Cependant, la recherche par son équipe de nouveaux moyens de traiter la LMA a été entravée par un écart subtil entre deux technologies utilisées pour comprendre la structure et la fonction des protéines – la cristallographie aux rayons X d’une part, et la microscopie électronique cryogénique (cryo-EM) d’autre part.

Reconstructions cryo-EM de KIX (rouge) pris en sandwich entre une enveloppe externe de MBP (violet) et une enveloppe interne d’apoferritine (bleu). La technique de prise en sandwich a permis aux chercheurs d’obtenir la meilleure image possible de KIX, une cible potentielle pour le traitement de la leucémie myéloïde aiguë. Crédit : Greg Stewart/SLAC National Accelerator Laboratory
Des chercheurs des écoles de médecine et d’ingénierie de l’université de Stanford et du SLAC National Accelerator Laboratory du ministère de l’énergie ont découvert un moyen de combler cette lacune en utilisant une cage moléculaire pour stabiliser certaines protéines de taille moyenne, ce qui permet de les imager pour la première fois avec la cryo-EM, qui peut révéler des détails au niveau presque atomique. Les premiers auteurs, Kaiming Zhang, boursier postdoctoral à Stanford, et Naoki Horikoshi, professeur adjoint invité, au moment des recherches, et ses collègues ont publié leurs résultats récemment dans la revue ACS Central Science.
Selon Soichi Wakatsuki, professeur au SLAC et à Stanford, le problème concerne KIX, une partie de la protéine de liaison CREB (CBP) que les cellules cancéreuses de la LMA utilisent pour transcrire des gènes importants pour la croissance et la survie. Si les chercheurs comprenaient mieux sa structure, ils pourraient concevoir des médicaments qui inhibent KIX et empêchent les cellules cancéreuses de se répliquer. Mais les efforts déployés pour étudier la protéine par cristallographie aux rayons X n’ont pas abouti : La taille relativement importante de la molécule – selon les normes de la cristallographie – la rend plus difficile à cristalliser, et même lorsqu’elle l’a été, les particularités de ce processus ont rendu plus difficile l’analyse des parties de KIX que les concepteurs de médicaments aimeraient cibler.
En même temps, la protéine KIX est un peu trop petite en soi pour être étudiée efficacement avec la cryo-EM. Pour obtenir de bonnes images d’une protéine avec la cryo-EM, explique Wakatsuki, il faut être capable de localiser de nombreuses copies de la protéine dans une image de microscope électronique, puis de déterminer comment elles sont orientées – si elles sont pliées dans un sens ou dans l’autre, etc. Ce n’est qu’en trouvant et en alignant de nombreuses images d’une protéine que les méthodes de cryo-EM permettent d’obtenir des structures à haute résolution. La taille relativement petite de KIX – par rapport aux normes de cryo-EM – rend cette tâche difficile. Une autre option, la résonance magnétique nucléaire, a été utilisée pour déterminer la structure de la protéine KIX lorsqu’elle est liée à d’autres molécules naturelles, mais cette méthode nécessite une préparation et une analyse approfondies. Elle est donc loin d’être idéale pour déterminer rapidement la structure des molécules et, par conséquent, pour étudier les effets d’éventuels médicaments inhibiteurs de la protéine KIX.
La solution est venue à Wakatsuki et Zhang – qui travaillait dans le laboratoire de Wah Chiu, professeur au SLAC et à Stanford – lors d’un déjeuner à Tokyo, où ils travaillaient sur un autre projet : Ils ont pris en sandwich des lots de protéines KIX entre une molécule centrale en forme de boule et une cage moléculaire extérieure. Cette “double enveloppe” étant beaucoup plus grande que les molécules KIX individuelles, il serait plus facile de la repérer et de l’orienter dans les images cryo-EM, ce qui faciliterait l’obtention d’images à haute résolution des molécules KIX elles-mêmes.
En plus d’observer la structure de KIX, Wakatsuki a déclaré que son laboratoire et celui de Chiu ont travaillé avec Sakamoto et Ron Dror, professeur d’informatique à Stanford, et ont pu ajouter d’autres molécules au mélange pour voir si elles pouvaient se lier à KIX et potentiellement inhiber sa fonction. L’équipe rapporte qu’elle a déjà réussi à rendre cette liaison environ 200 fois plus forte, ce qui pourrait aider les scientifiques…développer des médicaments qui sont efficaces à des doses plus faibles. “Le nom du jeu est de trouver des composés qui inhibent KIX à des concentrations plus faibles”, a déclaré Wakatsuki. “Ce n’est pas encore suffisant, mais nous avons fait des progrès”.
Les résultats de l’équipe suggèrent également que cette méthode pourrait s’avérer utile pour d’autres protéines de taille intermédiaire qui sont difficiles à étudier avec la cryo-EM ou la cristallographie aux rayons X – y compris, peut-être, certaines protéines virales. “Nous allons de l’avant pour étendre l’applicabilité de cette approche”, a déclaré Wakatsuki.
La recherche a été financée par les National Institutes of Health, la Pediatric Cancer Research Foundation, le Maternal Child Health Research Institute, l’Université de Stanford et la Leukemia and Lymphoma Society. Zhang a reçu un financement de démarrage de l’Université des sciences et de la technologie de Chine.
Référence : “Cryo-EM, Protein Engineering, and Simulation Enable the Development of Peptide Therapeutics against Acute Myeloid Leukemia” par Kaiming Zhang, Naoki Horikoshi, Shanshan Li, Alexander S. Powers, Mikhail A. Hameedi, Grigore D. Pintilie, Hee-Don Chae, Yousuf A. Khan, Carl-Mikael Suomivuori, Ron O. Dror, Kathleen M. Sakamoto, Wah Chiu et Soichi Wakatsuki, 7 février 2022, ACS Central Science.
DOI: 10.1021/acscentsci.1c01090