Une étude innovante de cartographie du cerveau révèle qu’un seul souvenir est stocké dans de nombreuses régions du cerveau connectées.
Une étude innovante de cartographie à l’échelle du cerveau montre que les “engrammes”, les ensembles de neurones codant pour un souvenir, sont largement distribués, y compris dans des régions dont on n’avait pas conscience auparavant.
Une nouvelle étude de MIT’s Picower Institute for Learning and Memory provides the most extensive and rigorous evidence yet that the mammalian brain retains a single memory across a broadly distributed, functionally integrated complex spanning many brain regions, rather than in just one or a few spots.
Memory research pioneer Richard Semon had predicted such a “unified engram complex” more than a century ago, but achieving the new study’s confirmation of his hypothesis required the application of multiple newly developed technologies. The researchers found and ranked dozens of previously unknown memory-related areas in the study, demonstrating that memory recall becomes more behaviorally powerful when multiple memory-storing regions are reactivated rather than just one.
“When talking about memory storage we all usually talk about the hippocampus or the cortex,” said co-lead and co-corresponding author Dheeraj Roy. He began the research while a graduate student in the RIKEN-MIT Laboratory for Neural Circuit Genetics at The Picower Institute led by senior author Susumu Tonegawa, Picower Professor in the Departments of Biology and Brain and Cognitive Sciences. “This study reflects the most comprehensive description of memory encoding cells, or memory ‘engrams,’ distributed across the brain, not just in the well-known memory regions. It basically provides the first rank-ordered list for high-probability engram regions. This list should lead to many future studies, which we are excited about, both in our labs and by other groups.”
In addition to Dheeraj Roy, who is now a McGovern Fellow in the Broad Institute of MIT and Harvard and the lab of MIT neuroscience Professor Guoping Feng, the study’s other lead authors are Young-Gyun Park, Minyoung Kim, Ying Zhang, and Sachie Ogawa.
Mapping Memory
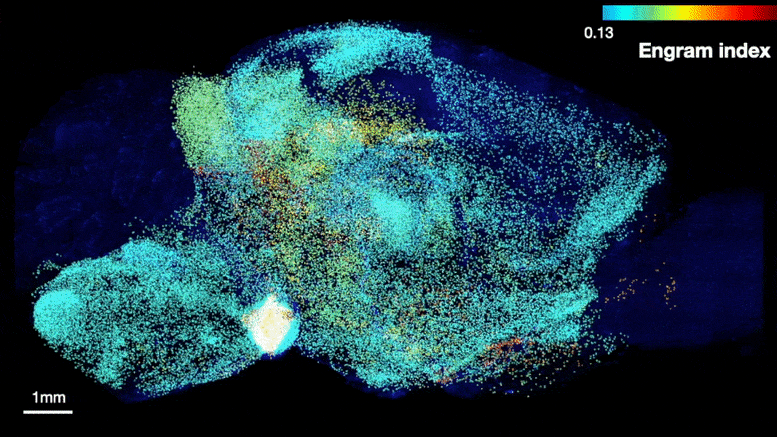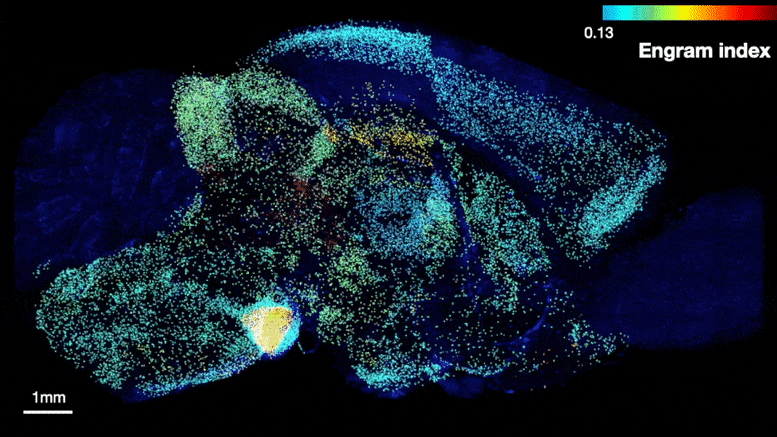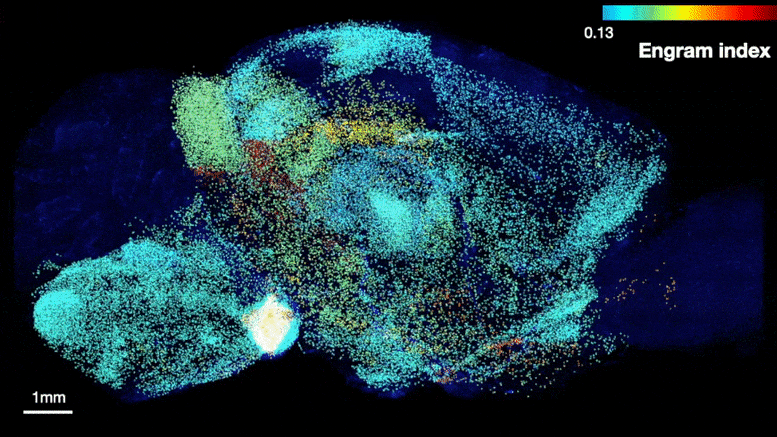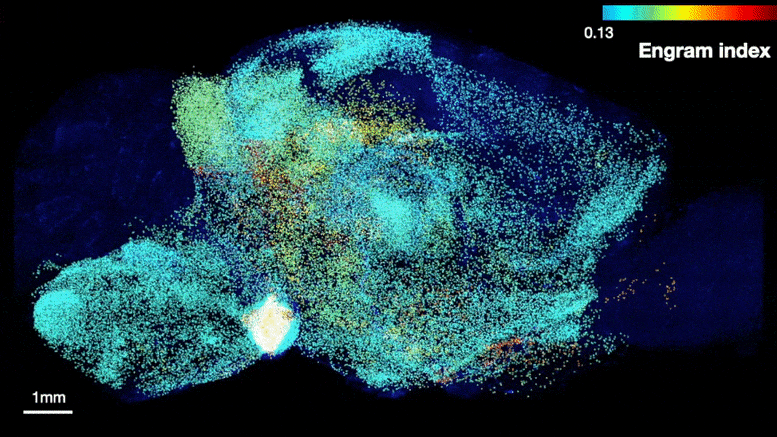
The team was able to map regions participating in an engram complex by conducting an unbiased analysis of more than 247 brain regions in mice who were taken from their home cage to another cage where they felt a small but memorable electrical zap. In one group of mice their neurons were engineered to become fluorescent when they expressed a gene required for memory encoding. In another group, cells activated by naturally recalling the zap memory (e.g. when the mice returned to the scene of the zap) were fluorescently labeled instead. Cells that were activated by memory encoding or by recall could therefore readily be seen under a microscope after the brains were preserved and optically cleared using a technology called SHIELD, developed by co-corresponding author Kwanghun Chung, Associate Professor in The Picower Institute, the Institute for Medical Engineering & Science and the Department of Chemical Engineering. By using a computer to count fluorescing cells in each sample, the team produced brain-wide maps of regions with apparently significant memory encoding or recall activity.
De nombreuses régions du cerveau susceptibles d’être impliquées dans l’encodage d’un souvenir (en haut) se sont également révélées être impliquées dans le rappel lors de la réactivation (en bas). Crédit : Laboratoire Tonegawa/Institut Picower du MIT
Les cartes ont mis en évidence de nombreuses régions censées participer à la mémoire, mais aussi de nombreuses autres qui ne le sont pas. Afin d’éliminer les régions qui auraient pu être activées par une activité sans rapport avec le souvenir du zappage, l’équipe a comparé ce qu’elle a observé chez les souris qui encodaient le zappage ou le rappel du zappage à ce qu’elle a observé dans le cerveau de souris témoins qui étaient simplement laissées dans leur cage d’origine. Cela leur a permis de calculer un “indice d’engramme” pour classer 117 régions du cerveau ayant une probabilité significative d’être impliquées dans le complexe d’engramme de mémoire. Ils ont approfondi l’analyse en créant de nouvelles souris dans lesquelles les neurones impliqués à la fois dans l’encodage et le rappel de la mémoire pouvaient être doublement marqués, révélant ainsi quelles cellules présentaient un chevauchement de ces activités.
Pour être réellement une cellule d’engramme, ont noté les auteurs, un neurone doit être activé à la fois dans l’encodage et le rappel.
“Ces expériences ont non seulement révélé une réactivation significative des engrammes dans les régions connues de l’hippocampe et de l’amygdale, mais également dans de nombreuses structures thalamiques, corticales, du mésencéphale et du tronc cérébral”, ont écrit les auteurs. “Fait important, lorsque nous avons comparé les régions du cerveau identifiées par l’analyse de l’indice d’engramme avec ces régions réactivées, nous avons observé que ~60% des régions étaient cohérentes entre les analyses.”
Manipulations de la mémoire
Après avoir classé les régions susceptibles d’être impliquées dans le complexe d’engramme, l’équipe s’est engagée dans plusieurs manipulations pour tester directement ses prédictions et pour déterminer comment les régions du complexe d’engramme pourraient travailler ensemble.
Par exemple, ils ont modifié des souris de façon à ce que les cellules activées par l’encodage de la mémoire puissent également être contrôlées par des flashs lumineux (une technique appelée “optogénétique”). Les chercheurs ont ensuite appliqué des flashs lumineux à des régions du cerveau sélectionnées dans leur liste d’index d’engrammes pour voir si la stimulation de ces régions pouvait reproduire artificiellement le comportement de gel sur place lié à la mémoire de la peur, même lorsque les souris étaient placées dans une cage “neutre” où le zap n’avait pas eu lieu.
“Il est frappant de constater que toutes ces régions du cerveau ont induit un rappel robuste de la mémoire lorsqu’elles ont été stimulées par optogénétique”, ont observé les chercheurs. De plus, la stimulation de régions qui, selon leur analyse, n’avaient pas d’importance pour la mémoire du zappage, n’a en fait produit aucun comportement de gel.
L’équipe a ensuite démontré comment les différentes régions d’un complexe d’engrammes sont connectées. Ils ont choisi deux régions bien connues de la mémoire, CA1 de l’hippocampe et l’amygdale basolatérale (BLA), et y ont activé par optogénétique des cellules engrammes pour induire un comportement de rappel de la mémoire dans une cage neutre. Ils ont constaté que la stimulation de ces régions produisait une activité de rappel de mémoire dans des zones spécifiques “en aval” identifiées comme étant des membres probables du complexe d’engramme. Parallèlement, l’inhibition optogénétique du rappel naturel de la mémoire par zap dans CA1 ou la BLA (c’est-à-dire lorsque les souris sont replacées dans la cage où elles ont subi le zap) a entraîné une réduction de l’activité dans les zones en aval du complexe engramme par rapport à ce qu’ils ont mesuré chez les souris dont le rappel naturel n’était pas entravé.
D’autres expériences ont montré que les réactivations optogénétiques des neurones du complexe d’engramme suivaient des schémas similaires à ceux observés lors du rappel naturel de la mémoire. Ayant établi que l’encodage et le rappel de la mémoire naturelle semblent se produire à travers un large complexe d’engrammes, l’équipe a décidé de tester si la réactivation de plusieurs régions améliorerait le rappel de la mémoire par rapport à la réactivation d’une seule. Après tout, des expériences antérieures ont montré que l’activation d’une seule zone d’engramme n’améliore pas la mémoire.ne produit pas un rappel aussi vif que le rappel naturel. Cette fois, l’équipe a utilisé un moyen chimique pour stimuler différentes régions du complexe d’engrammes et, ce faisant, elle a constaté que la stimulation simultanée de trois régions impliquées produisait un comportement de gel plus robuste que la stimulation d’une ou deux régions seulement.
Signification du stockage distribué
Roy a déclaré qu’en stockant un seul souvenir dans un complexe aussi étendu, le cerveau pourrait rendre la mémoire plus efficace et plus résistante.
“Différents engrammes de mémoire peuvent nous permettre de recréer des souvenirs plus efficacement lorsque nous essayons de nous souvenir d’un événement antérieur (et de même pour l’encodage initial où différents engrammes peuvent apporter des informations différentes de l’expérience originale)”, a-t-il déclaré. “Deuxièmement, dans les états pathologiques, si quelques régions sont altérées, les mémoires distribuées nous permettraient de nous souvenir d’événements antérieurs et, d’une certaine manière, seraient plus robustes face aux dommages régionaux.”
À long terme, cette deuxième idée pourrait suggérer une stratégie clinique pour traiter les troubles de la mémoire : “Si certains troubles de la mémoire sont dus à un dysfonctionnement de l’hippocampe ou du cortex, pourrions-nous cibler des cellules engrammes sous-étudiées dans d’autres régions, et une telle manipulation pourrait-elle rétablir certaines fonctions de la mémoire ?”
Ce n’est qu’une des nombreuses nouvelles questions que les chercheurs peuvent poser maintenant que l’étude a révélé une liste des endroits où chercher au moins un type de mémoire dans le cerveau des mammifères.
Référence : “Brain-wide mapping reveals that engrams for a single memory are distributed across multiple brain regions” par Dheeraj S. Roy, Young-Gyun Park, Minyoung E. Kim, Ying Zhang, Sachie K. Ogawa, Nicholas DiNapoli, Xinyi Gu, Jae H. Cho, Heejin Choi, Lee Kamentsky, Jared Martin, Olivia Mosto, Tomomi Aida, Kwanghun Chung et Susumu Tonegawa, 4 avril 2022, Nature Communications.
DOI: 10.1038/s41467-022-29384-4
Les autres auteurs de l’article sont Nicholas DiNapoli, Xinyi Gu, Jae Cho, Heejin Choi, Lee Kamentsky, Jared Martin, Olivia Mosto et Tomomi Aida.
Les sources de financement comprennent la Fondation JPB, le RIKEN Center for Brain Science, le Howard Hughes Medical Institute, un Warren Alpert Distinguished Scholar Award, les National Institutes of Health, le Burroughs Wellcome Fund, le Searle Scholars Program, un Packard Award in Science and Engineering, un NARSAD Young Investigator Award, le McKnight Foundation Technology Award, la NCSOFT Cultural Foundation et l’Institute for Basic Science.


