
Des chercheurs de l’UC San Diego introduisent la cellule intégrée à plusieurs échelles (MuSIC), une technique qui combine la microscopie, la biochimie et l’intelligence artificielle, révélant des composants cellulaires auparavant inconnus qui peuvent fournir de nouveaux indices sur le développement humain et la maladie. (Rendu conceptuel de l’artiste.) Crédit : UC San Diego Health Sciences
Une technique basée sur l’intelligence artificielle révèle des composants cellulaires auparavant inconnus qui peuvent fournir de nouveaux indices sur le développement humain et la maladie.
La plupart des maladies humaines peuvent être attribuées à des parties défectueuses d’une cellule – une tumeur est capable de se développer parce qu’un gène n’a pas été traduit avec précision en une protéine particulière ou une maladie métabolique survient parce que les mitochondries ne se déclenchent pas correctement, par exemple. Mais pour comprendre quelles parties d’une cellule peuvent mal tourner dans une maladie, les scientifiques doivent d’abord avoir une liste complète des parties.
En combinant la microscopie, les techniques de biochimie et l’intelligence artificielle, les chercheurs de la faculté de médecine de l’Université de Californie à San Diego et leurs collaborateurs ont fait ce qu’ils pensent être un grand pas en avant dans la compréhension des cellules humaines.
La technique, connue sous le nom de Multi-Scale Integrated Cell (MuSIC), est décrite le 24 novembre 2021 dans La nature.
« Si vous imaginez une cellule, vous imaginez probablement le diagramme coloré de votre manuel de biologie cellulaire, avec les mitochondries, le réticulum endoplasmique et le noyau. Mais est-ce toute l’histoire ? Certainement pas », a déclaré Trey Ideker, PhD, professeur à la faculté de médecine de l’UC San Diego et au Moores Cancer Center. « Les scientifiques ont compris depuis longtemps qu’il y a plus que nous ne savons pas, mais maintenant nous avons enfin un moyen d’aller plus loin. »
Ideker a dirigé l’étude avec Emma Lundberg, PhD, du KTH Royal Institute of Technology de Stockholm, en Suède, et de l’Université de Stanford.
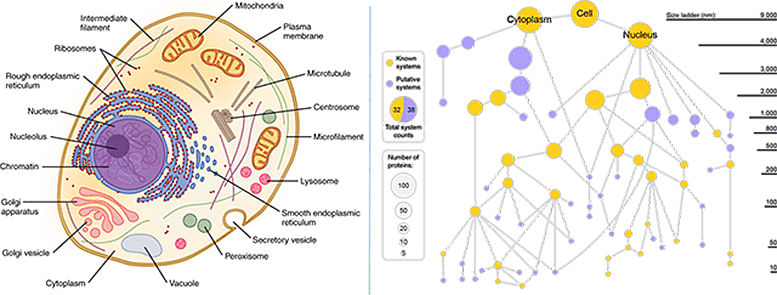
À gauche : les diagrammes de cellules de manuels classiques impliquent que toutes les parties sont clairement visibles et définies. (Crédit : OpenStax/Wikimedia). À droite : une nouvelle carte cellulaire générée par la technique MuSIC révèle de nombreux nouveaux composants. Les nœuds dorés représentent les composants cellulaires connus, les nœuds violets représentent les nouveaux composants. La taille du nœud reflète le nombre de protéines distinctes dans ce composant. Crédit: UC San Diego Health Sciences
Dans l’étude pilote, MuSIC a révélé environ 70 composants contenus dans une lignée cellulaire de rein humain, dont la moitié n’avait jamais été vue auparavant. Dans un exemple, les chercheurs ont repéré un groupe de protéines formant une structure inconnue. En travaillant avec Gene Yeo, PhD, collègue de l’UC San Diego, ils ont finalement déterminé que la structure était un nouveau complexe de protéines qui se lie ARN. Le complexe est probablement impliqué dans l’épissage, un événement cellulaire important qui permet la traduction des gènes en protéines et aide à déterminer quels gènes sont activés à quel moment.
L’intérieur des cellules – et les nombreuses protéines qui s’y trouvent – sont généralement étudiés à l’aide de l’une des deux techniques suivantes : l’imagerie au microscope ou l’association biophysique. Avec l’imagerie, les chercheurs ajoutent des balises fluorescentes de différentes couleurs aux protéines d’intérêt et suivent leurs mouvements et associations dans le champ de vision du microscope. Pour examiner les associations biophysiques, les chercheurs peuvent utiliser un anticorps spécifique à une protéine pour la retirer de la cellule et voir ce qui y est attaché.
L’équipe s’intéresse depuis de nombreuses années à la cartographie du fonctionnement interne des cellules. Ce qui est différent avec MuSIC, c’est l’utilisation de l’apprentissage en profondeur pour cartographier la cellule directement à partir d’images de microscopie cellulaire.
« La combinaison de ces technologies est unique et puissante car c’est la première fois que des mesures à des échelles très différentes sont réunies », a déclaré le premier auteur de l’étude, Yue Qin, étudiant diplômé en bioinformatique et biologie des systèmes au laboratoire d’Ideker.
Les microscopes permettent aux scientifiques de voir jusqu’au niveau d’un seul micron, soit à peu près la taille de certains organites, comme les mitochondries. Les éléments plus petits, tels que les protéines individuelles et les complexes protéiques, ne peuvent pas être vus au microscope. Les techniques de biochimie, qui partent d’une seule protéine, permettent aux scientifiques de descendre à l’échelle nanométrique. (Un nanomètre est un milliardième de mètre, ou 1 000 microns.)
« Mais comment comblez-vous cet écart entre l’échelle du nanomètre et celle du micron ? Cela a longtemps été un gros obstacle dans les sciences biologiques », a déclaré Ideker, qui est également fondateur de l’UC Cancer Cell Map Initiative et du Centre de biologie computationnelle et de bioinformatique de l’UC San Diego. “Il s’avère que vous pouvez le faire avec l’intelligence artificielle – en examinant les données de plusieurs sources et en demandant au système de les assembler dans un modèle de cellule.”
L’équipe a formé la plate-forme d’intelligence artificielle MuSIC pour examiner toutes les données et construire un modèle de la cellule. Le système ne mappe pas encore le contenu des cellules à des emplacements spécifiques, comme un diagramme de manuel, en partie parce que leurs emplacements ne sont pas nécessairement fixes. Au lieu de cela, les emplacements des composants sont fluides et changent en fonction du type de cellule et de la situation.
Ideker a noté qu’il s’agissait d’une étude pilote pour tester MuSIC. Ils n’ont examiné que 661 protéines et un seul type de cellule.
“La prochaine étape claire est de souffler à travers toute la cellule humaine”, a déclaré Ideker, “et ensuite de passer à différents types de cellules, personnes et espèces. À terme, nous pourrons peut-être mieux comprendre la base moléculaire de nombreuses maladies en comparant les différences entre les cellules saines et malades.
Référence : « A multi-scale map of cell structure fusing protein images and interactions » par Yue Qin, Edward L. Huttlin, Casper F. Winsnes, Maya L. Gosztyla, Ludivine Wacheul, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, Anna Bäckström, Laura Pontano Vaites, John J. Lee, Wei Ouyang, Sophie N. Liu, Tian Zhang, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg, Steven P. Gygi, Jianzhu Ma, J. Wade Harper, Gene W. Yeo, Denis LJ Lafontaine, Emma Lundberg et Trey Ideker, 24 novembre 2021, La nature.
DOI : 10.1038 / s41586-021-04115-9
Les co-auteurs incluent : Maya L. Gosztyla, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, John J. Lee, Sophie N. Liu, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg, UC San Diego ; Edward L. Huttlin, Laura Pontano Vaites, Tian Zhang, Steven P. Gygi, J. Wade Harper, Harvard Medical School; Casper F. Winsnes, Anna Bäckström, Wei Ouyang, KTH Royal Institute of Technology ; Ludivine Wacheul, Denis LJ Lafontaine, Université Libre de Bruxelles ; et Jianzhu Ma, Université de Pékin.
Le financement de cette recherche est venu, en partie, des National Institutes of Health (subventions U54CA209891, U01MH115747, F99CA264422, P41GM103504, R01HG009979, U24HG006673, U41HG009889, R01HL137223, R01HG004659, R50CA243885), Google Ventures Foundation, Alice, Erling Wallenberg Foundation (subvention 2016.0204), Swedish Research Council (subvention 2017-05327), Fonds belge de la Recherche Scientifique, Université Libre de Bruxelles, European Joint Program on Rare Diseases, Région Wallonne, Internationale Brachet Stiftung et Epitran COST action (subvention CA16120 ).
Divulgations : Trey Ideker est co-fondateur de, membre du conseil consultatif scientifique et détient une participation dans Data4Cure, Inc. Ideker est également membre du conseil consultatif scientifique, détient une participation et reçoit un financement de recherche parrainé par Ideaya BioSciences, Inc. Gene Yeo est co-fondateur, membre du conseil d’administration, du conseil consultatif scientifique, actionnaire et consultant rémunéré pour Locanabio et Eclipse BioInnovations. Yeo est également professeur invité à l’Université nationale de Singapour. Les termes de ces accords ont été examinés et approuvés par l’Université de Californie à San Diego conformément à ses politiques en matière de conflits d’intérêts. Emma Lundberg est membre des conseils consultatifs scientifiques et détient des participations dans Cartography Biosciences, Nautilus Biotechnology et Interline Therapeutics. J. Wade Harper est co-fondateur de, membre du conseil consultatif scientifique et détient une participation dans Caraway Therapeutics. Harper est également conseiller scientifique fondateur pour Interline Therapeutics.



