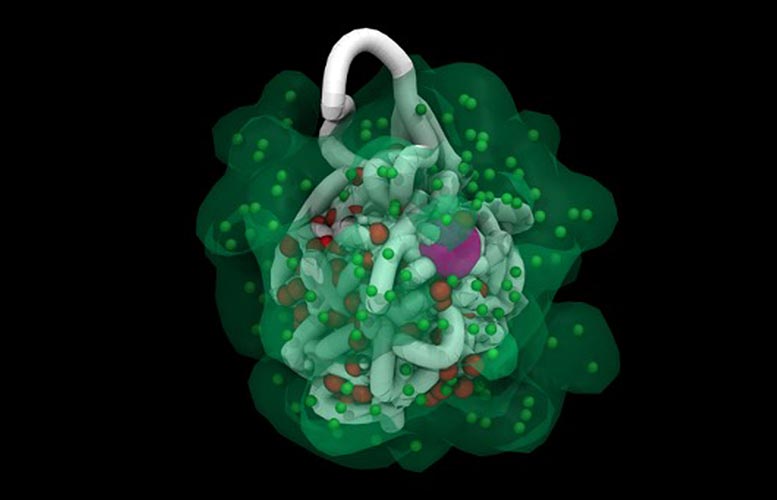
Des particules d’ARN envahissent un chromosome X d’une souris dans une nouvelle visualisation de l’inactivation du chromosome X. Crédit : Laboratoire national de Los Alamos
La combinaison des données de laboratoire avec la puissance du supercalcul révèle le rôle de ARN et la structure des chromosomes dans la régulation de l’expression des gènes.
En utilisant une modélisation dynamique pilotée par un superordinateur et basée sur des données expérimentales, les chercheurs peuvent désormais sonder le processus qui désactive un chromosome X dans les embryons de mammifères femelles. Cette nouvelle capacité aide les biologistes à comprendre le rôle de l’ARN et de la structure du chromosome dans le processus d’inactivation du X, menant à une compréhension plus approfondie de l’expression des gènes et ouvrant de nouvelles voies vers des traitements médicamenteux pour les troubles et les maladies génétiques.
“C’est la première fois que nous avons été en mesure de modéliser tout l’ARN qui se propage autour du chromosome et de l’arrêter”, a déclaré Anna Lappala, scientifique invitée au Laboratoire national de Los Alamos et physicienne des polymères au Massachusetts General Hospital et au département de Harvard. de biologie moléculaire. Lappala est le premier auteur de l’article publié le 4 octobre 2021 dans le Actes de l’Académie nationale des sciences. « À partir des seules données expérimentales, qui sont en 2D et statiques, vous n’avez pas la résolution de voir un chromosome entier à ce niveau de détail. Avec cette modélisation, nous pouvons voir les processus régulant l’expression des gènes, et la modélisation est fondée sur les données expérimentales 2D de nos collaborateurs du Massachusetts General Hospital et de Harvard.
Le modèle, considéré comme 4D car il montre le mouvement, y compris le temps comme quatrième dimension, fonctionne sur les superordinateurs de Los Alamos. Le modèle intègre également des données expérimentales de génomes de souris obtenues grâce à une méthode moléculaire appelée 4DHiC. La méthodologie moléculaire et informatique combinée est une première.
Dans la visualisation, les particules d’ARN pullulent sur le chromosome X. Les brins enchevêtrés ressemblant à des spaghettis se tordent, changent de forme, puis les particules s’engouffrent et pénètrent dans les profondeurs du chromosome, l’éteignant. Voir la visualisation ici :
“La méthode nous permet de développer un modèle interactif de ce processus épigénétique”, a déclaré Jeannie T. Lee, professeur de génétique à la Harvard Medical School et vice-présidente en biologie moléculaire au Massachusetts General Hospital, dont le laboratoire a fourni les données expérimentales qui sous-tendent le modèle.
L’épigénétique est l’étude des changements dans l’expression des gènes et des traits héréditaires qui n’impliquent pas de mutations dans le génome.
“Ce qui manquait sur le terrain, c’est un moyen pour un utilisateur qui n’est pas calé en informatique d’entrer de manière interactive dans un chromosome”, a déclaré Lee. Elle a comparé l’utilisation du modèle de Los Alamos à l’utilisation de Google Earth, où « vous pouvez zoomer sur n’importe quel emplacement sur un chromosome X, choisir votre gène préféré, voir les autres gènes qui l’entourent et voir comment ils interagissent ». Cette capacité pourrait donner un aperçu de la propagation des maladies, par exemple, a-t-elle déclaré.
Sur la base des travaux de cet article, Los Alamos développe actuellement un navigateur de style Google Earth où tout scientifique peut télécharger ses données génomiques et les visualiser dynamiquement en 3D à divers grossissements, a déclaré Karissa Sanbonmatsu, biologiste structurale au Laboratoire national de Los Alamos, auteur correspondant de l’article et chef de projet dans le développement de la méthode de calcul.
Chez les mammifères, un embryon femelle est conçu avec deux chromosomes X, un hérité de chaque parent. L’inactivation de X arrête le chromosome, une étape cruciale pour la survie de l’embryon, et les variations de l’inactivation de X peuvent déclencher une variété de troubles du développement.
Le nouveau modèle de Los Alamos facilitera une compréhension plus approfondie de l’expression des gènes et des problèmes connexes, ce qui pourrait conduire à des traitements pharmacologiques pour diverses maladies et troubles génétiques, a déclaré Lee.
“Notre objectif principal était de voir le chromosome changer de forme et de voir les niveaux d’expression des gènes au fil du temps”, a déclaré Sanbonmatsu.
Pour comprendre comment les gènes sont activés et désactivés, a déclaré Sanbonmatsu, « cela aide vraiment à connaître la structure du chromosome. L’hypothèse est qu’un chromosome compacté et étroitement structuré a tendance à désactiver les gènes, mais il n’y a pas beaucoup d’armes à feu à ce sujet. En modélisant des structures 3D en mouvement, nous pouvons nous rapprocher de la relation entre le compactage structurel et la désactivation des gènes. »
Lee a comparé la structure du chromosome à l’origami. Une forme compliquée semblable à une grue en origami offre beaucoup de surface pour l’expression des gènes et pourrait être biologiquement préférée pour rester active.
Le modèle montre une variété de sous-structures dans le chromosome. Quand il est fermé, “c’est un processus au coup par coup dans lequel certaines sous-structures sont conservées mais certaines sont dissoutes”, a déclaré Sanbonmatsu. «Nous voyons des étapes de début, intermédiaire et finale, à travers une transition graduelle. C’est important pour l’épigénétique car c’est la première fois que nous avons pu analyser la transition structurelle détaillée dans un changement épigénétique.
La modélisation montre également des gènes à la surface du chromosome qui échappent à l’inactivation du chromosome X, confirmant les premiers travaux expérimentaux. Dans le modèle, ils se regroupent et interagissent ou travaillent apparemment ensemble à la surface du chromosome.
Dans un autre aperçu de la modélisation, « Alors que le chromosome passe d’un X actif, quand il est encore assez grand, à un X inactif compact, c’est-à-dire plus petit, nous remarquons qu’il y a un noyau du chromosome qui est extrêmement dense, mais la surface est beaucoup moins dense. Nous voyons aussi beaucoup plus de mouvement à la surface », a déclaré Lappala. “Ensuite, il y a une région intermédiaire qui n’est ni trop rapide ni trop lente, où le chromosome peut se réarranger.”
Un X inactif peut s’activer plus tard dans un processus appelé activation liée à l’âge du X inactif. “Il est associé à des problèmes dans les cellules sanguines en particulier qui sont connus pour provoquer une auto-immunité”, a déclaré Lee. « Certaines recherches tentent d’activer pharmacologiquement le X inactif pour traiter les troubles neurologiques chez les enfants en leur redonnant quelque chose qui manque sur leur chromosome X actif. Par exemple, un enfant pourrait avoir une mutation qui peut causer une maladie. Nous pensons que si nous pouvons réactiver la copie normale sur le X inactif, nous aurions alors un traitement épigénétique pour cette mutation. »
Référence : « 4D chromosome reconstruction elucidates the spatial reorganization of the mammalian X-chromosome » par Anna Lappala, Chen Yu Wang, Andrea Kriz, Hunter Michalk, Kevin Tan, Jeannie Lee et Karissa Sanbonmatsu, 4 octobre 2021, Actes de l’Académie nationale des sciences.
Financement : Programme de recherche et développement dirigé par un laboratoire au Laboratoire national de Los Alamos.