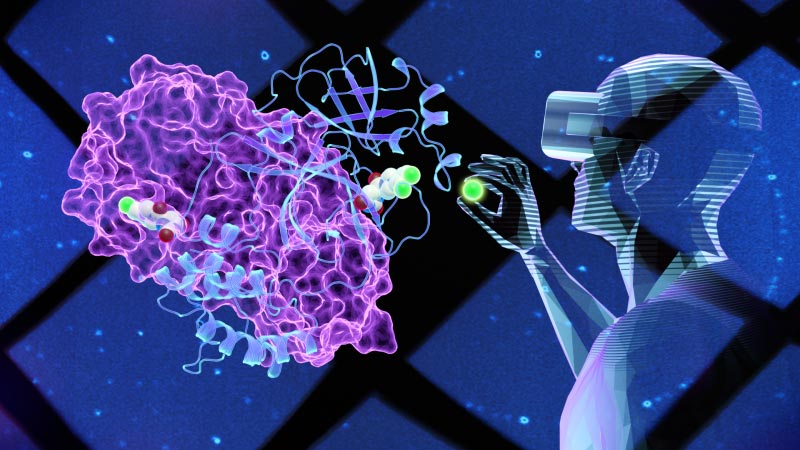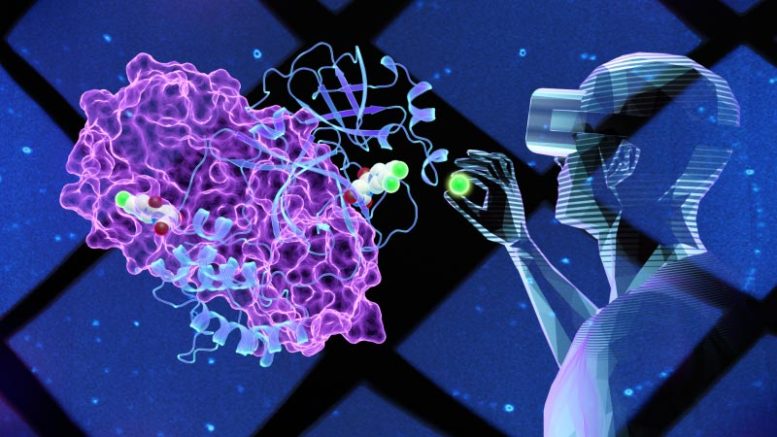
La technologie de réalité virtuelle a permis aux scientifiques de regarder «à l’intérieur» du virus Covid-19 et de développer une nouvelle molécule capable d’inhiber sa principale enzyme protéase. Crédit : Jill Hemman/ORNL
La technologie de réalité virtuelle (VR) permet aux scientifiques de créer des modèles 3D d’un objet, puis d’aller virtuellement « à l’intérieur » pour regarder autour de lui afin de mieux comprendre sa structure et sa fonction.
C’est ce que les chercheurs du laboratoire national d’Oak Ridge (ORNL) du ministère de l’Énergie (DOE) ont fait pour étudier la SRAS-CoV-2 virus qui a causé la COVID-19[feminine pandémie. L’équipe a utilisé des neutrons et des rayons X pour cartographier une partie de la structure interne du coronavirus afin de créer un modèle 3D précis. Plus précisément, les scientifiques ont cartographié la protéase principale (Mpro), une enzyme impliquée dans la réplication du virus, à laquelle ils avaient ajouté une petite molécule préliminaire découverte à l’aide d’un criblage informatique à grande vitesse.
En utilisant la réalité virtuelle pour examiner le modèle enzymatique, les scientifiques ont pratiquement construit différentes petites molécules en modifiant leurs structures pour voir si des composés nouvellement conçus pouvaient s’adapter ou se lier à un site clé sur la surface de l’enzyme Mpro. Une liaison suffisamment forte pourrait inhiber ou bloquer le fonctionnement de l’enzyme, ce qui est vital pour empêcher le virus de se multiplier chez les patients atteints de COVID-19.
Pour déterminer les effets de modifications chimiques spécifiques sur la manière dont les 19 candidats inhibiteurs se lient à l’enzyme Mpro, l’équipe a synthétisé chaque molécule d’inhibiteur et mesuré leurs forces de liaison. Plus la liaison est forte, plus l’inhibiteur bloquerait efficacement le fonctionnement de l’enzyme et la réplication du virus.
L’un des inhibiteurs testés, étiqueté HL-3-68, a démontré une capacité supérieure à se lier à et à inhiber la fonction de Mpro par rapport aux autres qui ont été testés. Les détails de l’étude, intitulée « Déterminants structurels, électroniques et électrostatiques de la liaison des inhibiteurs aux sous-sites S1 et S2 dans la protéase principale du SARS-CoV-2 », sont publiés dans le Journal de chimie médicinale.
“Notre étude a été conçue pour mieux comprendre comment les molécules se lient au site actif de l’enzyme Mpro, qui joue un rôle clé dans la réplication du SARS-CoV-2”, a déclaré l’auteur principal Daniel Kneller. “En testant les molécules que nous avons conçues, nous en avons découvert une contenant un seul chlore supplémentaire atome qui a montré une plus grande capacité à inhiber Mpro. Cette nouvelle structure chimique est différente de ce qui a été précédemment étudié par la communauté mondiale et pourrait ouvrir de nouvelles voies de recherche avec des possibilités passionnantes pour lutter contre le SRAS-CoV-2. »
Le site actif de l’enzyme Mpro est commun à d’autres types de coronavirus et ne semble pas muter facilement, ce qui représente une opportunité de concevoir éventuellement un traitement antiviral qui fonctionne contre plusieurs variantes du SRAS-CoV-2 et d’autres coronavirus.
Il est tout aussi important que le site actif soit différent de ceux connus dans les enzymes humaines, ce qui minimiserait le potentiel de liaison involontaire pouvant entraîner des effets secondaires chez les patients.
Les mesures aux rayons X et la production des échantillons d’enzyme Mpro ont été effectuées par le Centre de biologie structurale et moléculaire à l’aide des installations de la source de neutrons de spallation (SNS) de l’ORNL et des ressources du réacteur isotopique à haut flux (HFIR). Les candidats inhibiteurs ont été synthétisés par les co-auteurs Hui Li et Peter Bonnesen du groupe Nanomatériaux macromoléculaires du Center for Nanophase Materials Sciences (CNMS).
«Cette étude a combiné une pléthore de méthodes biophysiques, biochimiques et de biologie moléculaire, et comprenait une analyse structurelle assistée par réalité virtuelle et la construction de petites molécules, réunissant des scientifiques de l’ORNL, du laboratoire national d’Argonne, des instituts nationaux de la santé et de l’université du Tennessee. -Knoxville. La nature collaborative de l’étude nous a permis de découvrir les règles auxquelles les inhibiteurs de petites molécules doivent obéir lorsqu’ils se lient à l’enzyme afin d’être utiles pour les étapes ultérieures du long processus de conception et de développement de médicaments », a déclaré l’auteur correspondant Andrey Kovalevsky.
L’auteur co-correspondant Peter Bonnesen a ajouté : « Il s’agissait d’un nouveau projet passionnant sur lequel le CNMS devait travailler, et il s’appuyait sur notre expertise dans la synthèse de molécules organiques personnalisées pour nos utilisateurs. Pour ce projet, nous avons fourni à nos collègues SNS quelques molécules candidates à la fois. Au fur et à mesure que les résultats concernant l’efficacité des molécules en tant qu’inhibiteurs revenaient, l’équipe discuterait des ajustements à apporter à la structure moléculaire. Ensuite, Hui et moi retournions au laboratoire pour fabriquer ces nouveaux inhibiteurs candidats. »
L’étude a également mis en lumière la capacité de l’enzyme Mpro à changer de forme et à changer sa charge électrique de positive à négative, ou de négative à positive, selon la taille et la structure de la molécule inhibitrice à laquelle elle se lie. Ces caractéristiques sont importantes à comprendre lors du développement d’une molécule inhibitrice efficace.
Pour la recherche sur la diffusion des neutrons, les scientifiques ont utilisé le diffractomètre macromoléculaire à neutrons (MaNDi) du SNS pour sa capacité à collecter des données à partir des échantillons relativement petits avec lesquels l’équipe a dû travailler.
“En raison de la nature urgente de la recherche liée au virus SARS-CoV-2, nous n’avons pu cultiver que des échantillons relativement petits de l’enzyme Mpro”, a déclaré le co-auteur Leighton Coates. « Comme les échantillons plus petits diffusent faiblement les neutrons, ce qui entraîne des données neutroniques « bruyantes », l’analyse des données peut être difficile. La structure temporelle du faisceau de neutrons de l’instrument MaNDi nous a permis d’éliminer la majeure partie du bruit, augmentant ainsi le rapport signal sur bruit, nous donnant ainsi des données beaucoup plus utiles avec lesquelles travailler.
Les prochaines étapes pour les chercheurs de l’ORNL incluent le test des modifications chimiques de l’inhibiteur HL-3-68 pour déterminer si des composés nouvellement conçus peuvent se lier encore mieux que HL-3-68 pour inhiber plus efficacement l’enzyme Mpro et finalement empêcher le coronavirus de se répliquer.
Pendant ce temps, les chercheurs ont rendu leurs données accessibles au public via le Banque de données sur les protéines pour accélérer l’information et l’assistance des communautés scientifiques et médicales du monde. Bien sûr, davantage de recherches et de tests sont nécessaires pour valider l’efficacité et l’innocuité de tout inhibiteur en tant que traitement COVID-19. Cependant, cette étude pourrait offrir une opportunité à d’autres scientifiques de mener des recherches supplémentaires qui profiteraient à des milliards de personnes dans le monde.
Référence : « Déterminants structurels, électroniques et électrostatiques pour la liaison des inhibiteurs aux sous-sites S1 et S2 dans la protéase principale du SRAS-CoV-2 » par Daniel W. Kneller, Hui Li, Stephanie Galanie, Gwyndalyn Phillips, Audrey Labbé, Kevin L. Weiss, Qiu Zhang, Mark A. Arnould, Austin Clyde, Heng Ma, Arvind Ramanathan, Colleen B. Jonsson, Martha S. Head, Leighton Coates, John M. Louis, Peter V. Bonnesen et Andrey Kovalevsky, 27 octobre 2021, Journal de chimie médicinale.
DOI : 10.1021/acs.jmedchem.1c01475
Les autres co-auteurs de l’article sont Stephanie Galanie, Gwyndalyn Phillips, Audrey Labbé, Kevin L. Weiss, Qiu Zhang, Mark A. Arnould, Austin Clyde, Heng Ma, Arvind Ramanathan, Colleen B. Jonsson, Martha S. Head et John M. Louis. Hugh O’Neill de l’ORNL a assisté à la préparation des échantillons.
La recherche sur le COVID-19 à l’ORNL a été soutenue en partie par le Laboratoire national virtuel de biotechnologie de l’Office of Science, un consortium de laboratoires nationaux du DOE axé sur la réponse au COVID-19, avec un financement fourni par la loi Coronavirus CARES. Ce travail a également été soutenu par l’Institut national du diabète et des maladies digestives et rénales des National Institutes of Health.