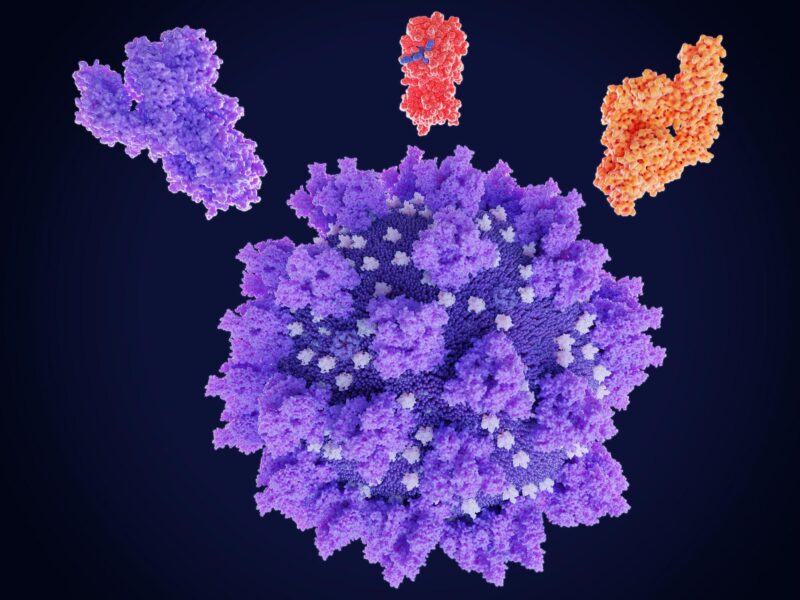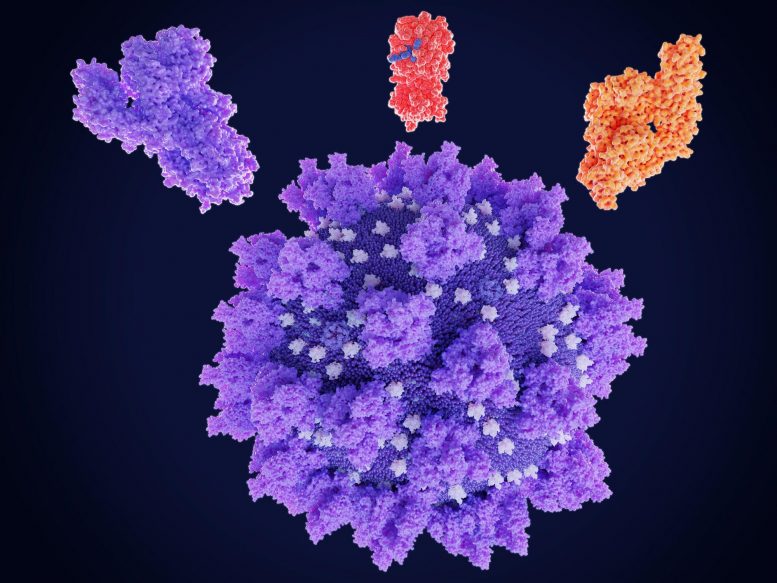
Des scientifiques de l’université associée à Liverpool ont montré à quel point SRAS-CoV-2 les protéases de type virus attaquent la cellule hôte particulière, et comment cela peut être ciblé pour arrêter la réplication du virus dans la culture cellulaire avec les médicaments actuellement utilisés en clinique ou même en cours de développement.
Les nouvelles découvertes, publiées aujourd’hui (21 septembre 2021) dans Communication marketing nature , offrent une ressource efficace pour comprendre la protéolyse dans le contexte associé à l’infection virale, et également pour éclairer le développement de stratégies ciblées pour inhiber l’herpès qui cause Covid-19.
Le SRAS-CoV-2 a été responsable de plus de 227 millions d’infections, et plus par rapport aux 4,6 millions de décès dans le monde tout au long de la pandémie. Les efforts pour vérifier, traiter et vacciner contre le virus bénéficient presque tous d’une meilleure connaissance de la biologie de base associée au SRAS-CoV-2.
Chaque protéase virale et mobile joue un rôle important dans la réplication du SARS-CoV-2, et les bloqueurs ciblant les protéases ont déjà réussi à inhiber le SARS-CoV-2 dans les versions de culture cellulaire.
Dans cette recherche, dirigée par l’Université ou le collège de Liverpool ainsi que l’Institut Pasteur de Paris, les chercheurs ont utilisé une approche de spectrométrie de masse pour étudier les événements de clivage protéolytique tout au long de l’infection par le SRAS-CoV-2.
“Les méthodes basées sur la spectrométrie de masse pour identifier les substrats de protéase existent depuis plusieurs années, mais elles n’ont vu qu’une application restreinte à la recherche de substrats viraux et n’avaient pas déjà été appliquées à la recherche de protéolyse lors d’une infection à coronavirus”, explique le plomb. écrivain Dr. Emmott Edward Cullen, Autre tenure-Track à l’Université Start of Systems, Molecular and Integrative Le domaine de la biologie.
Le groupe a découvert des sites de clivage non identifiés auparavant dans plusieurs protéines virales saines, y compris les principales protéines antigéniques S plus N, qui sont les principales cibles des efforts de dépistage des injections et des anticorps.
Ils découvriront des augmentations significatives des événements de seins cellulaires compatibles avec les seins par la protéase majeure du SRAS-CoV-2 (Mpro) et ont identifié 14 substrats de haute confiance possibles à partir des protéases principales et de type papaïne, validant le sous-ensemble avec in vitro dosages.
Ils ont continué à montrer que l’épuisement des siRNA de ces protéines mobiles inhibe la réplication du SRAS-CoV-2, et que les médicaments ciblant deux de ces protéines : la tyrosine kinase SRC plus Ser/Thr kinase MYLK, ont montré une réduction dose-dépendante du SRAS CoV-2. titres.
Chaque Bafetinib (un médicament anticancéreux frais) plus Sorafenib (un médicament autorisé utilisé pour traiter le cancer des organes du rein et du foie) a montré une inhibition du SRAS-CoV-2 à des niveaux qui n’ont pas conduit à une cytotoxicité dans un type d’infection de lignée cellulaire humaine.
Le docteur Emmott a déclaré : « Une meilleure compréhension de la manière dont les seins protéolytiques sont régulés, modulent l’activité des protéines et favorisent la réplication virale va être cruciale pour se concentrer sur les substrats cellulaires associés aux protéases virales comme une stratégie thérapeutique.
« À mesure que d’autres variantes du SRAS-CoV-2 émergent, l’incorporation particulière d’informations sur la modification traductionnelle des articles de blog provenant d’études comme celle-ci peut également soutenir les tentatives de prédire les phénotypes à partir des informations génétiques sur les versions émergentes. ”
Point de référence : « La caractérisation de la protéolyse tout au long de l’infection par le SRAS-CoV-2 reconnaît les sites Web de clivage viral et les foyers cellulaires ayant un potentiel thérapeutique » par Bjoern Meyer, Jeanne Chiaravalli, Stacy Gellenoncourt, Philip Brownridge, Dominic P. Bryne, Leonard A. Daly, Arturas Grauslys , Marius Walter, Fabrice Agou, Lisa A. Chakrabarti, Charles S. Craik, Claire E. Eyers, Patrick A. Eyers, Yann Gambin, Toby R. Jones, Emma Sierecki, Eric Verdin, Marco Vignuzzi plus Edward Emmott, 21 septembre 2021, Communication marketing nature .
DOI : 10. 1038/s41467-021-25796-w