Par

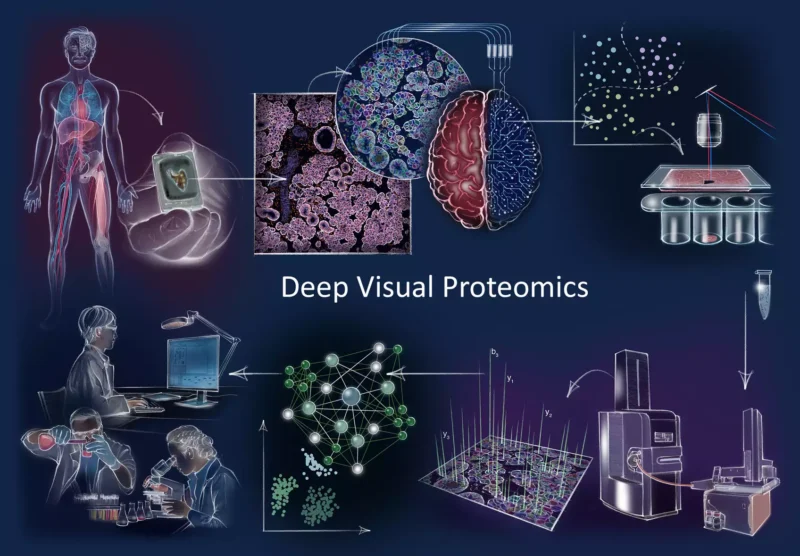
Concept et flux de travail de la protéomique visuelle profonde Dans le sens des aiguilles d’une montre : Deep Visual Proteomics (DVP) combine l’imagerie à haute résolution, l’analyse d’images guidée par l’intelligence artificielle (IA) pour la classification et l’isolement de cellules uniques avec un nouveau flux de travail protéomique ultra-sensible. La DVP associe une imagerie riche en données de cultures cellulaires ou de tissus archivés de biobanques de patients à une segmentation cellulaire basée sur l’apprentissage profond et à une identification des types et des états cellulaires basée sur l’apprentissage automatique. Les objets cellulaires ou subcellulaires d’intérêt classés par l’IA (non) supervisée subissent une microdissection laser automatisée et un profilage protéomique basé sur la spectrométrie de masse (MS). L’analyse ultérieure des données bioinformatiques permet l’exploration de données pour découvrir des signatures protéiques fournissant des informations moléculaires sur la variation du protéome dans les états de santé et de maladie au niveau des cellules individuelles. Crédit : MPI de biochimie
Comment certains patients développent-ils une résistance au traitement du cancer ? La nouvelle méthode connue sous le nom de “Deep Visual Proteomics” pourrait aider les médecins à se rapprocher de la réponse et à identifier les vulnérabilités des tumeurs cancéreuses.
Il n’est jamais facile pour les médecins de comprendre pourquoi certaines maladies se développent dans notre corps. La vieillesse, les habitudes à risque comme le tabagisme et la génétique peuvent tous jouer un rôle.
Cependant, les causes exactes et spécifiques de maladies graves comme le cancer restent inconnues.
Aujourd’hui, une méthode révolutionnaire connue sous le nom de “Deep Visual Proteomics” pourrait contribuer à changer cette situation. Une équipe internationale de chercheurs dirigée par l’université de Copenhague a créé cette technique, qui a récemment été appliquée aux cellules cancéreuses dans une nouvelle étude publiée dans la revue scientifique de premier plan . Nature Biotechnology.
“Notre nouveau concept, la protéomique visuelle profonde, pourrait changer la donne en matière de pathologie moléculaire dans les hôpitaux. Avec cette méthode, nous pouvons identifier des milliers de protéines et déterminer leur nombre”, explique Andreas Mund, premier auteur de la nouvelle étude.
“Pour ce faire, nous prenons un échantillon de tissu et analysons uniquement les cellules tumorales qu’il contient. Cette ‘liste’ de protéines est appelée protéome. Ces protéomes révèlent les mécanismes qui régissent le développement des tumeurs et exposent directement de nouvelles cibles thérapeutiques à partir d’une seule tranche de tissu d’une biopsie de patient cancéreux. Il expose un cosmos de molécules à l’intérieur de ces cellules cancéreuses”, explique Andreas Mund, professeur associé au Centre de recherche sur les protéines de la Fondation Novo Nordisk (CPR) et membre de l’équipe du professeur Matthias Mann qui a mené ce développement au CPR et à l’Institut Max Planck de biochimie.
Important pour les services de pathologie
La raison pour laquelle les chercheurs s’intéressent tant aux protéines est qu’elles sont en fait certaines des pièces les plus importantes du puzzle de presque toutes les maladies. Les protéines sont communément appelées les “chevaux de trait de la cellule”.
“Lorsque quelque chose ne va pas à l’intérieur de nos cellules et que nous tombons malades, vous pouvez être sûr que les protéines sont impliquées de nombreuses manières différentes. C’est pourquoi la cartographie du paysage protéique peut nous aider à déterminer pourquoi une tumeur peut se développer chez un patient particulier, quelles sont les vulnérabilités de cette tumeur, et aussi quelle stratégie de traitement pourrait s’avérer la plus bénéfique”, explique Matthias Mann, professeur.
Dans la nouvelle étude, les chercheurs ont appliqué la “Deep Visual Proteomics” à des cellules provenant de patients atteints de carcinome à cellules acineuses et de mélanome. Cette étude a été réalisée en collaboration avec des chercheurs de l’hôpital universitaire de Zélande, à Roskilde.
“Cette méthode unique combine l’architecture des tissus avec l’expression de milliers de protéines spécifiques aux cellules sélectionnées. Elle permet aux chercheurs d’étudier les interactions entre les cellules cancéreuses et les cellules environnantes, avec des implications majeures pour le futur traitement clinique du cancer. Récemment, nous avons diagnostiqué un cas clinique très complexe avec 2 composantes différentes et les résultats de l’analyse DVP”, explique Lise Mette Rahbek Gjerdrum, consultante et professeur associé de recherche clinique au département de pathologie de l’hôpital universitaire de Zélande et au département de médecine clinique de l’université de Copenhague.
Pathologie numérique, apprentissage profond, microscopie et spectrométrie de masse.
Deep Visual Proteomics intègre les avancées de quatre technologies différentes en un seul flux de travail. Tout d’abord, la microscopie avancée génère des cartes tissulaires à haute résolution. Ensuite, des algorithmes d’apprentissage automatique sont utilisés pour classer les cellules avec précision avant les microdissections au laser et la collecte de cellules uniques. Ensuite, seules les cellules normales ou malades d’un type particulier sont analysées par spectroscopie de masse, ce qui permet de cartographier les protéines.et la compréhension des mécanismes de la santé et de la maladie.
“Grâce à cette technologie, nous pouvons effectivement relier les caractéristiques physiologiques des cellules vues au microscope aux fonctions des protéines. Cela n’était pas possible auparavant et nous sommes convaincus que cette méthode peut être appliquée à d’autres maladies, et pas seulement au cancer”, déclare Andreas Mund.
Référence : “Deep Visual Proteomics définit l’identité et l’hétérogénéité de la cellule unique” par Andreas Mund, Fabian Coscia, András Kriston, Réka Hollandi, Ferenc Kovács, Andreas-David Brunner, Ede Migh, Lisa Schweizer, Alberto Santos, Michael Bzorek, Soraya Naimy, Lise Mette Rahbek-Gjerdrum, Beatrice Dyring-Andersen, Jutta Bulkescher, Claudia Lukas, Mark Adam Eckert, Ernst Lengyel, Christian Gnann, Emma Lundberg, Peter Horvath et Matthias Mann, 19 mai 2022,Nature Biotechnology.
DOI: 10.1038/s41587-022-01302-5