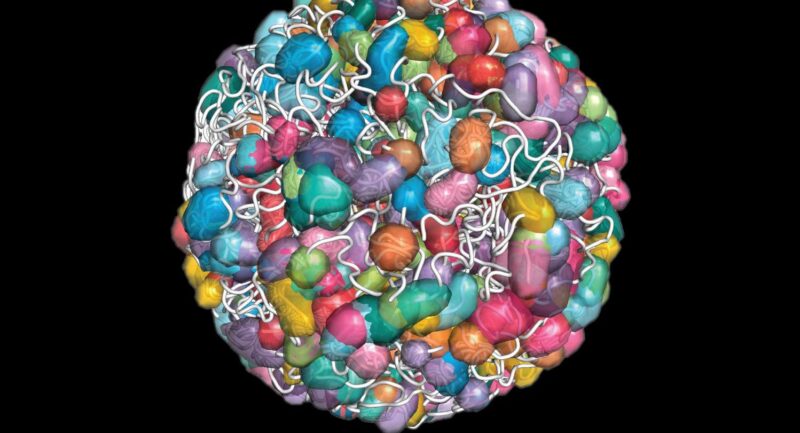
L’ADN et la machinerie cellulaire associée sont rassemblés et organisés par certains ARN non codants, chaque région étant représentée sous une forme de bonbon de couleur différente dans ce modèle. Crédit : Avec l’aimable autorisation d’Inna-Marie Strazhnik
Le génome humain peut être considéré comme une bibliothèque massive, contenant plus de 20 000 « manuels d’instructions » différents : vos gènes. Par exemple, il existe des gènes qui contiennent des informations pour construire une cellule du cerveau, une cellule de la peau, un globule blanc, etc. Il existe même des gènes qui contiennent des informations sur la régulation du génome lui-même, comme des livres qui expliquent comment organiser une bibliothèque. La capacité à réguler l’expression des gènes— en d’autres termes, la capacité de la cellule à activer ou désactiver diverses constellations de gènes — est à la base de la raison pour laquelle différentes cellules (telles qu’une cellule musculaire ou une cellule cérébrale) ont des formes et des fonctions différentes.
Pour qu’une bibliothèque soit utile à un lecteur, elle doit être organisée de manière facilement consultable. Par exemple, tous les livres relatifs à l’histoire du monde peuvent être sur une seule étagère, tandis que les livres de cuisine peuvent être dans une section entièrement différente de la bibliothèque. Dans un noyau cellulaire, il y a plus de 1,80 mètre de matériel génétique emballé dans un espace 50 fois plus petit que la largeur d’un cheveu humain. Comment la « bibliothèque » du noyau est-elle organisée ? Lorsqu’une cellule a besoin de réguler certains gènes, comment la machinerie cellulaire trouve-t-elle les bons parmi 20 000 autres ?
Un nouvel article du laboratoire de Mitchell Guttman, professeur de biologie, utilise un nouvel outil puissant qui peut scruter le monde du matériel génétique de la cellule (ADN et ARN) afin de trouver des réponses à ces questions.
Dirigé par l’ancienne étudiante diplômée du laboratoire Guttman, Sofia Quinodoz (PhD ’20), maintenant boursière postdoctorale Hanna Gray à université de Princeton— l’équipe a découvert que les molécules d’ARN non codant sont responsables de l’établissement de « compartiments » dans le noyau et du guidage des molécules clés vers des régions précises du génome. Les ARN non codants sont des molécules qui ne codent pas pour les protéines et qui ont plutôt un éventail de fonctions qui sont souvent encore mystérieuses pour les biologistes. Dans l’analogie de la bibliothèque, les molécules d’ARN non codantes agissent comme des « étagères » qui organisent différents groupes de gènes et la machinerie qui interagit avec eux.
Comprendre comment le matériel génétique est organisé spatialement est un élément crucial pour comprendre le fonctionnement de base de la vie. Le dysfonctionnement au sein du noyau est une caractéristique de nombreuses maladies, y compris le cancer, les troubles neurodégénératifs et autres.
La recherche a été rendue possible grâce à un outil puissant développé dans le laboratoire Guttman qui permet des vues détaillées du monde de l’ARN, appelé RD-SPRITE (RNA and DNA Split-Pool Recognition of Interactions by Tag Extension). Essentiellement, RD-SPRITE fonctionne en marquant des molécules d’ARN et d’ADN avec de minuscules codes-barres uniques en fonction de leur emplacement ; l’analyse des codes-barres peut alors vous dire quelles molécules étaient à quelles positions dans la cellule.
« Cet outil est quelque chose dont je rêvais depuis que je suis étudiant diplômé. C’est remarquable que Sofia ait pu y arriver », déclare Guttman. «Cela change ce que nous pouvons voir dans le monde de l’ARN. C’est comme développer un nouveau microscope ; vous pouvez commencer à regarder des choses que vous ne pouviez jamais voir auparavant. Cette découverte sur l’ARN et l’organisation est la pointe de l’iceberg en termes de ce que nous pouvons commencer à trouver dans ces données. »
L’équipe prévoit d’utiliser RD-SPRITE pour comparer l’organisation spatiale du noyau entre les cellules saines et les types de cellules malades, afin de comprendre comment l’expression des gènes et la structure physique du noyau peuvent être affectées dans les états pathologiques.
Référence : « L’ARN favorise la formation de compartiments spatiaux dans le noyau » par Sofia A. Quinodoz, Joanna W. Jachowicz, Prashant Bhat, Noah Ollikainen, Abhik K. Banerjee, Isabel N. Goronzy, Mario R. Blanco, Peter Chovanec, Amy Chow, Yolanda Markaki, Jasmine Thai, Kathrin Plath et Mitchell Guttman, 4 novembre 2021, Cellule.
DOI : 10.1016/j.cell.2021.10.014
L’article est intitulé “L’ARN favorise la formation de compartiments spatiaux dans le noyau”. Sofia Quinodoz est la première auteure de l’article, ainsi que la co-seconde auteure de la chercheuse postdoctorale Caltech Joanna Jachowicz, l’étudiante diplômée Prashant Bhat et l’ancienne chercheuse postdoctorale Noah Ollikainen. En plus de Guttman, les autres coauteurs incluent l’ancien étudiant diplômé Abhik Banerjee, l’étudiante diplômée Isabel Goronzy, le chercheur scientifique Mario Blanco, l’ancien chercheur postdoctoral Peter Chovanec, la chercheuse scientifique principale Amy Chow, Yolanda Markaki de l’UCLA, l’ancienne assistante technique de recherche Jasmine Thai et Kathrin Plath de l’UCLA. Le financement a été fourni par le Howard Hughes Medical Institute, la National Science Foundation, les National Institutes of Health, le programme de formation des scientifiques médicaux de l’UCLA-Caltech, l’American Cancer Society, la Division of Biology and Biological Engineering de Caltech, le National Heart Lung et l’Institut du sang et le USC Programme MD/PhD.