Votre DNA holds the blueprint to build your body, but it’s a living document: Adjustments to the design can be made by epigenetic marks.
Epigenetic marks are modifications to DNA bases that don’t change the underlying genetic code, but “write” extra information on top of it that can be inherited along with your genome. Epigenetic marks usually regulate gene expression — turn genes on or off — particularly during early development or when your body is under stress. They can also suppress “jumping genes” — transposable elements that threaten the integrity of your genome.
In humans and other eukaryotes, two principal epigenetic marks are known. A team from the Marine Biological Laboratory (MBL) has discovered a third, novel epigenetic mark – one formerly known only in bacteria — in bdelloid rotifers, small freshwater animals. This fundamental and surprising discovery is reported this week in Nature Communications.
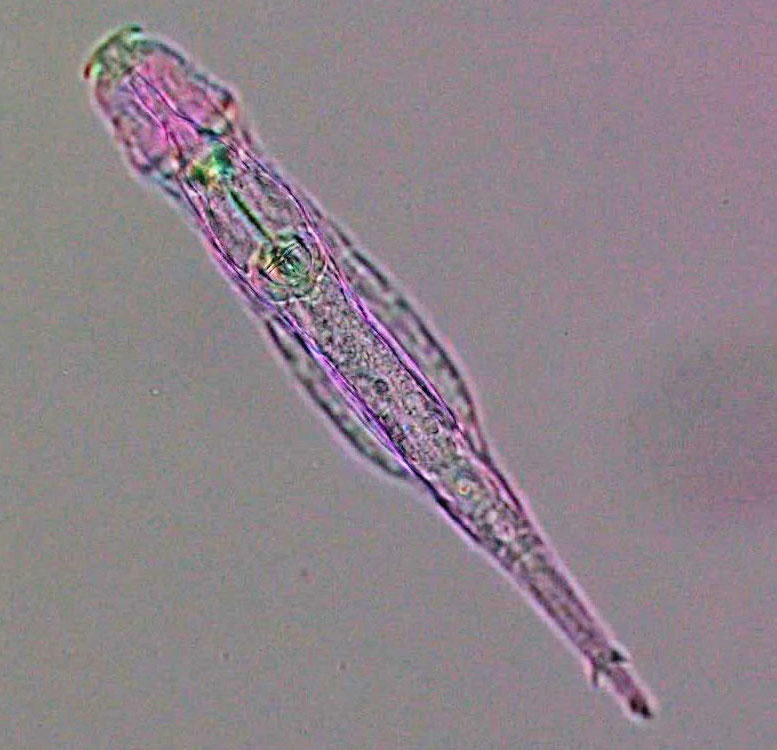
Bdelloid rotifer (Adineta vaga) under polychromatic polarization microscope. Credit: M. Shribak and I. Yushenova
“We discovered back in 2008 that bdelloid rotifers are very good at capturing foreign genes,” said senior author Irina Arkhipova, senior scientist in the MBL’s Josephine Bay Paul Center. “What we’ve found here is that rotifers, about 60 million years ago, accidentally captured a bacterial gene that allowed them to introduce a new epigenetic mark that was not there before.” This is the first time that a horizontally transferred gene has been shown to reshape the gene regulatory system in a eukaryote.
“This is very unusual and has not been previously reported,” Arkhipova said. “Horizontally transferred genes are thought to preferentially be operational genes, not regulatory genes. It is hard to imagine how a single, horizontally transferred gene would form a new regulatory system, because the existing regulatory systems are already very complicated.”
“It’s almost unbelievable,” said co-first author Irina Yushenova, a research scientist in Arkhipova’s lab. “Just try to picture, somewhere back in time, a piece of bacterial DNA happened to be fused to a piece of eukaryotic DNA. Both of them became joined in the rotifer’s genome and they formed a functional enzyme. That’s not so easy to do, even in the lab, and it happened naturally. And then this composite enzyme created this amazing regulatory system, and bdelloid rotifers were able to start using it to control all these jumping transposons. It’s like magic.”
Un rotifère bdelloïde (Adineta vaga) en train de se nourrir sous un microscope à polarisation polychromatique. Crédit : M. Shribak et I. Yushenova
“Vous ne voulez pas que les transposons sautent partout dans votre génome”, a déclaré le premier auteur Fernando Rodriguez, également chercheur dans le laboratoire d’Arkhipova. “Ils vont tout gâcher et il faut donc les garder sous contrôle. Et le système épigénétique qui permet d’accomplir cela est différent chez les différents animaux. Dans ce cas, un transfert horizontal de gènes de la bactérie vers les rotifères bdelloïdes a créé un nouveau système épigénétique chez les animaux qui n’a pas été décrit auparavant.”
“Les rotifères bdelloïdes, en particulier, doivent garder leurs transposons sous contrôle car ils se reproduisent principalement de manière asexuée”, a déclaré Arkhipova. “Les lignées asexuées disposent de moins de moyens pour supprimer la prolifération des transposons délétères, de sorte que l’ajout d’une couche supplémentaire de protection pourrait empêcher un effondrement mutationnel. En effet, la teneur en transposons est beaucoup plus faible chez les bdelloïdes que chez les eucaryotes sexuels qui ne disposent pas de cette couche épigénétique supplémentaire dans leur système de défense du génome.”
Dans les deux marques épigénétiques précédemment connues chez les eucaryotes, un groupe méthyle est ajouté à une base de l’ADN, soit la cytosine ou l’adénine. La marque nouvellement découverte par l’équipe est également une modification de la cytosine, mais avec un positionnement du groupe méthyle distinct, de type bactérien – récapitulant essentiellement les événements évolutifs d’il y a plus de deux milliards d’années, lorsque les marques épigénétiques conventionnelles des premiers eucaryotes sont apparues.
Les rotifères bdelloïdes sont des animaux extrêmement résistants, comme l’ont découvert au fil des ans les laboratoires Arkhipova et David Mark Welch au MBL. Ils peuvent se dessécher complètement (dessiccation) pendant des semaines ou des mois, puis revenir à la vie lorsque l’eau devient disponible. Pendant leurs phases de dessiccation, leur ADN se brise en de nombreux morceaux. “Lorsqu’ils se réhydratent ou rendent les extrémités de leur ADN accessibles, cela peut être l’occasion pour des fragments d’ADN étrangers provenant de bactéries, de champignons ou de microalgues ingérés de se transférer dans le génome du rotifère”, a déclaré Arkhipova. Les chercheurs ont découvert qu’environ 10 % du génome des rotifères provient de sources non métazoaires.
Pourtant, le laboratoire d’Arkhipova a été surpris de trouver un gène dans le génome du rotifère qui ressemblait à une méthyltransférase bactérienne (une méthyltransférase catalyse le transfert d’un groupe méthyle à l’ADN). “Nous avons émis l’hypothèse que ce gène conférait cette nouvelle fonction de suppression des transposons, et nous avons passé les six dernières années à prouver que c’était bien le cas”, a déclaré Arkhipova.
Il est trop tôt pour savoir quelles pourraient être les implications de la découverte de ce nouveau système épigénétique chez les rotifères. “Une bonne comparaison est le système CRISPR-Cas chez les bactéries, qui a commencé comme une découverte de recherche fondamentale. Aujourd’hui, CRISPR-Cas9 est utilisé partout comme outil d’édition de gènes dans d’autres organismes”, a déclaré Rodriguez. “Il s’agit d’un nouveau système. Aura-t-il des applications, des implications pour la recherche future ? C’est difficile à dire.”
Ces découvertes ouvrent la porte à de nouveaux outils et à de nouvelles directions de recherche pour étudier la fonction et la résilience du génome dans ce système de rotifères. À l’avenir, ces connaissances pourraient être appliquées de manière créative pour avoir un impact sur la société en cette période de changements environnementaux rapides.
Référence : “Bacterial N4-methylcytosine as an epigenetic mark in eukaryotic DNA” par Fernando Rodriguez, Irina A. Yushenova, Daniel DiCorpo et Irina R. Arkhipova, 28 février 2022, Nature Communications.
DOI: 10.1038/s41467-022-28471-w