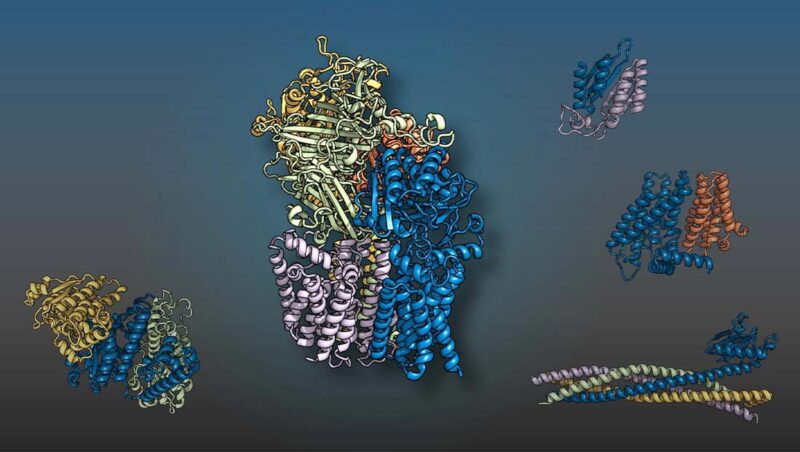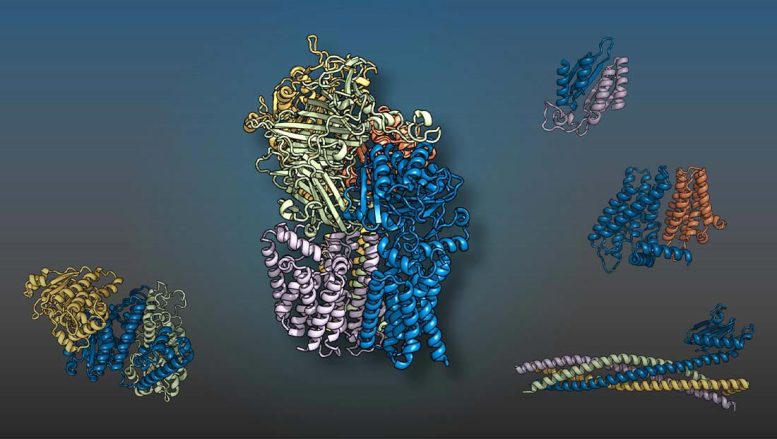
Les protéines de levure présentées dans différentes couleurs se réunissent sous forme de complexes à deux, trois, quatre et cinq membres comme des pièces de puzzle 3D pour exécuter des fonctions cellulaires. Une équipe internationale dirigée par des chercheurs de l’UT Southwestern et de l’Université de Washington a prédit les structures à l’aide de techniques d’intelligence artificielle. Crédit: UT Southwestern Medical Center
Les recherches menées par UT Southwestern et le Université de Washington pourrait conduire à une multitude de cibles médicamenteuses.
Des chercheurs de l’UT Southwestern et de l’Université de Washington ont dirigé une équipe internationale qui a utilisé l’intelligence artificielle (IA) et l’analyse évolutive pour produire des modèles 3D d’interactions protéiques eucaryotes. L’étude, publiée dans Science, a identifié pour la première fois plus de 100 complexes protéiques probables et a fourni des modèles structurels pour plus de 700 complexes jusqu’alors non caractérisés. Des informations sur la manière dont les paires ou les groupes de protéines s’assemblent pour effectuer des processus cellulaires pourraient conduire à une multitude de nouvelles cibles médicamenteuses.
“Nos résultats représentent une avancée significative dans la nouvelle ère de la biologie structurelle dans laquelle le calcul joue un rôle fondamental”, a déclaré Qian Cong, Ph.D., professeur adjoint au Eugene McDermott Center for Human Growth and Development avec un poste secondaire en biophysique. .
Qian Cong, Ph.D. Crédit: UT Southwestern Medical Center
Le Dr Cong a dirigé l’étude avec David Baker, Ph.D., professeur de biochimie et mentor postdoctoral du Dr Cong à l’Université de Washington avant son recrutement à l’UT Southwestern. L’étude compte quatre coauteurs principaux, dont le biologiste computationnel de l’UT Southwestern Jimin Pei, Ph.D.
Les protéines fonctionnent souvent en paires ou en groupes appelés complexes pour accomplir toutes les tâches nécessaires pour maintenir un organisme en vie, a expliqué le Dr Cong. Alors que certaines de ces interactions sont bien étudiées, beaucoup restent un mystère. Construire des interactomes complets – ou des descriptions de l’ensemble complet des interactions moléculaires dans une cellule – éclairerait de nombreux aspects fondamentaux de la biologie et donnerait aux chercheurs un nouveau point de départ pour développer des médicaments qui encouragent ou découragent ces interactions. Le Dr Cong travaille dans le domaine émergent de l’interactomique, qui combine la bioinformatique et la biologie.
Jusqu’à récemment, un obstacle majeur à la construction d’un interactome était l’incertitude sur les structures de nombreuses protéines, un problème que les scientifiques tentent de résoudre depuis un demi-siècle. En 2020 et 2021, une société appelée DeepMind et le laboratoire du Dr Baker ont publié indépendamment deux technologies d’IA appelées AlphaFold (AF) et RoseTTAFold (RF) qui utilisent différentes stratégies pour prédire les structures des protéines en fonction des séquences des gènes qui les produisent.
Dans la présente étude, le Dr Cong, le Dr Baker et leurs collègues ont développé ces outils de prédiction de structure d’IA en modélisant de nombreux complexes protéiques de levure. La levure est un organisme modèle commun pour les études biologiques fondamentales. Pour trouver des protéines susceptibles d’interagir, les scientifiques ont d’abord recherché dans les génomes de champignons apparentés des gènes ayant acquis des mutations de manière liée. Ils ont ensuite utilisé les deux technologies d’IA pour déterminer si ces protéines pouvaient être assemblées dans des structures 3D.
Leurs travaux ont identifié 1 505 complexes protéiques probables. Parmi ceux-ci, 699 avaient déjà été caractérisés structurellement, vérifiant l’utilité de leur méthode. Cependant, il n’y avait que des données expérimentales limitées soutenant 700 des interactions prédites, et 106 autres n’avaient jamais été décrites.
Pour mieux comprendre ces complexes mal caractérisés ou inconnus, les équipes de l’Université de Washington et de l’UT Southwestern ont travaillé avec des collègues du monde entier qui étudiaient déjà ces protéines ou des protéines similaires. En combinant les modèles 3D que les scientifiques de la présente étude avaient générés avec les informations des collaborateurs, les équipes ont pu acquérir de nouvelles connaissances sur les complexes protéiques impliqués dans la maintenance et le traitement de l’information génétique, la construction cellulaire et les systèmes de transport, le métabolisme, ADN réparation, et d’autres domaines. Ils ont également identifié des rôles pour des protéines dont les fonctions étaient auparavant inconnues sur la base de leurs interactions nouvellement identifiées avec d’autres protéines bien caractérisées.
“Les travaux décrits dans notre nouvel article préparent le terrain pour des études similaires sur l’interactome humain et pourraient éventuellement aider à développer de nouveaux traitements pour les maladies humaines”, a ajouté le Dr Cong.
Le Dr Cong a noté que les structures complexes de protéines prédites générées dans cette étude peuvent être téléchargées à partir de ModèleArchive. Ces structures et d’autres générées à l’aide de cette technologie dans les études futures seront une riche source de questions de recherche pour les années à venir, a-t-elle déclaré.
Référence : « Structures calculées des complexes de protéines eucaryotes de base » par Ian R. Humphreys, Jimin Pei, Minkyung Baek, Aditya Krishnakumar, Ivan Anishchenko, Sergey Ovchinnikov, Jing Zhang, Travis J. Ness, Sudeep Banjade, Saket R. Bagde, Viktoriya G Stancheva, Xiao-Han Li, Kaixian Liu, Zhi Zheng, Daniel J. Barrero, Upasana Roy, Jochen Kuper, Israel S. Fernández, Barnabas Szakal, Dana Branzei, Josep Rizo, Caroline Kisker, Eric C. Greene, Sue Biggins, Scott Keeney, Elizabeth A. Miller, J. Christopher Fromme, Tamara L. Hendrickson, Qian Cong et David Baker, 11 novembre 2021, Science.
DOI : 10.1126/science.abm4805
Le Dr Cong est un boursier de la Southwestern Medical Foundation en recherche biomédicale. Parmi les autres chercheurs de l’UTSW qui ont contribué à cette étude, citons Jing Zhang et Josep Rizo, Ph.D., titulaire de la chaire Virginia Lazenby O’Hara en biochimie.
Les institutions collaboratrices comprennent : l’Université Harvard, l’Université Wayne State, l’Université Cornell, le Laboratoire de biologie moléculaire du MRC, le Memorial Sloan Kettering Cancer Center, la Gerstner Sloan Kettering Graduate School of Biomedical Sciences, le Fred Hutchinson Cancer Research Center, Université de Columbia, Université de Würzburg en Allemagne, Hôpital de recherche pour enfants St Jude, Institut FIRC d’oncologie moléculaire à Milan, Italie, et Conseil national de recherche, Institut de génétique moléculaire à Rome, Italie.
Ce travail a été soutenu par la Southwestern Medical Foundation, le Cancer Prevention and Research Institute of Texas (CPRIT) (RP210041), Amgen, Microsoft, la Washington Research Foundation, Howard Hughes Medical Institute, National Science Foundation (DBI 1937533), National Institutes of Health (R35GM118026, R01CA221858, R35GM136258, R21AI156595), UK Medical Research Council (MRC_UP_1201/10), HHMI Gilliam Fellowship, Deutsche Forschungsgemeinschaft (KI-562/11-1, KI-562/7-1), chercheur de l’AIRC et Research Council Consolidator (IG23710 et 682190), Defence Threat Reduction Agency (HDTRA1-21-1-0007) et National Energy Research Scientific Computing Center.