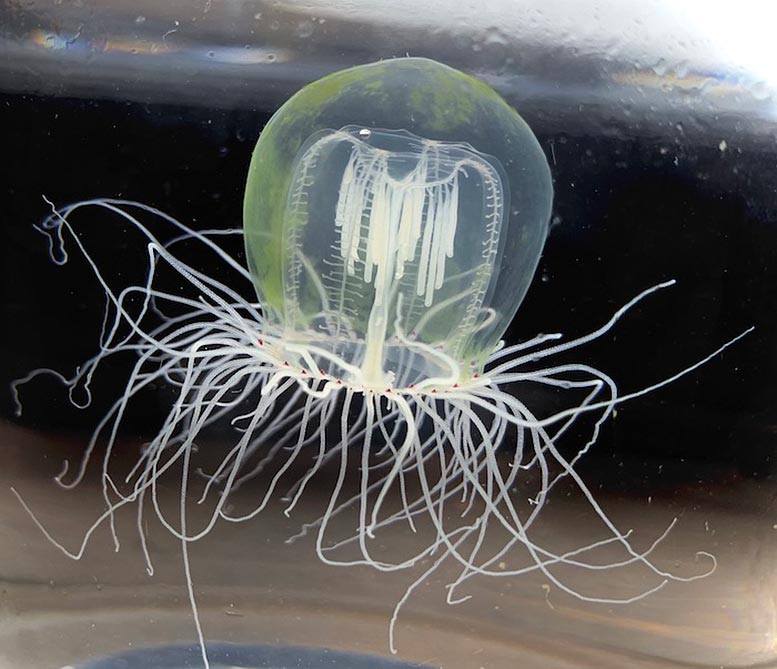
Cette minuscule méduse appelée Polyorchis pencillatus est connue sous le nom de méduse aux yeux rouges. Ses multiples yeux, appelés ocelles, sont visibles à la base des tentacules et contiennent des cellules sensibles à la lumière et un pigment rouge. Crédit : Anna Klompen
Certaines méduses ont des yeux simples, d’autres des yeux complexes. D’autres méduses n’ont pas d’yeux du tout. En effet, des recherches récentes ont montré que les yeux des méduses de différentes espèces ont évolué séparément et indépendamment de nombreuses fois et de différentes manières au cours de plusieurs millénaires, ce qui en fait un modèle idéal pour mieux comprendre comment ce trait s’exprime génétiquement.
Aujourd’hui, une équipe de chercheurs comprenant Paulyn Cartwright, professeur d’écologie et de biologie de l’évolution à l’Université du Kansas, Maria Pia Miglietta de l’Université du Texas A&M, Galveston, et le chercheur principal Todd Oakley de l’Université de Californie, Santa Barbara, a reçu une subvention de la National Science Foundation pour étudier comment la “convergence” des yeux de méduses – des événements répétés dans l’histoire de la vie – fournit une fenêtre sur le fonctionnement de l’évolution aux niveaux génétique, cellulaire et morphologique.
La part de la KU dans la subvention de la NSF s’élève à 494 890 dollars.
“Les yeux ont évolué plusieurs fois indépendamment chez les méduses”, a déclaré Cartwright. “Nous savons depuis un certain temps qu’il n’y a pas une seule origine des yeux chez tous les animaux, mais nous avons été surpris par le nombre de fois où ils ont évolué indépendamment chez les méduses.”
Maintenant, Cartwright et ses collaborateurs prévoient une “plongée profonde” scientifique dans les modèles d’évolution pour découvrir si les méduses utilisent les mêmes aspects ou des aspects différents de leur boîte à outils génétique pour construire les yeux chaque fois qu’elles ont évolué.
Les chercheurs prévoient de reconstruire un arbre phylogénomique détaillé des Medusozoa (un clade du phylum Cnidaria) pour retracer l’histoire de l’évolution des yeux des méduses et d’utiliser des transcriptomes comparatifs et des cellules uniques RNA sequencing to determine similarities and differences in genetic pathways of convergent eyes.
“Jellyfish are a really nice system for this, because they have a diversity of eyes, ranging from simple clusters of light-sensing cells to very complex eyes — camera-type eyes that can form images and have a lens, a cornea and a retina — so superficially, they can look identical to vertebrates,” Cartwright said.
In her KU lab, she plans to analyze many species of jellyfish to determine all genes expressed in single cells of different jellyfish eyes and find what’s shared among different instances of jellyfish and what’s changed in their basic genetic building blocks.
“Cells themselves have their own characteristics, and they’re really an outcome of many, many different genes being expressed — so sometimes we might miss an overall pattern by looking at individual genes,” the KU researcher said. “But if we look at all the genes that are expressed at the cell and what that particular outcome is, that might give us a different level of information. That’s why it’s great to look at all those different levels and see what is similar and what’s changed to really help us understand this very complicated question. Jellyfish are a great system to do this because they’re so amenable to these types of experiments. We can look at individual genes and how they’re expressed; we can look wholesale at all the genes that are expressed in those cells; and then we can see what is similar at the morphological level between those and what’s different.”
Part of the work under the new NSF grant will include travel to Panama, a hotspot of jellyfish biodiversity, to collect specimens. Cartwright and her colleagues will use the fruits of their research to construct a more detailed phylogenetic tree, or evolutionary history, of jellyfish.
“Jellyfish are very diverse — there’s a few thousand species,” Cartwright said. “Uncovering their exact evolutionary history has been really challenging in part because a lot of this diversification happened over half a billion years ago. The other challenge is to sample these organisms. Many of them live in open-ocean environments in the deep sea, and some of them are incredibly small and hard to find. So, we’re looking at ancient divergence times within a diverse and difficult-to-sample group. This has been a challenge that I’ve been working on for at least the last 20 years of my career, so we’re really excited in the age of genomics because getting more data and being able to sequence more genes and throwing more DNA sequences at this problem is expected to be very promising at resolving some of these relationships amongst Cnidarians.”


