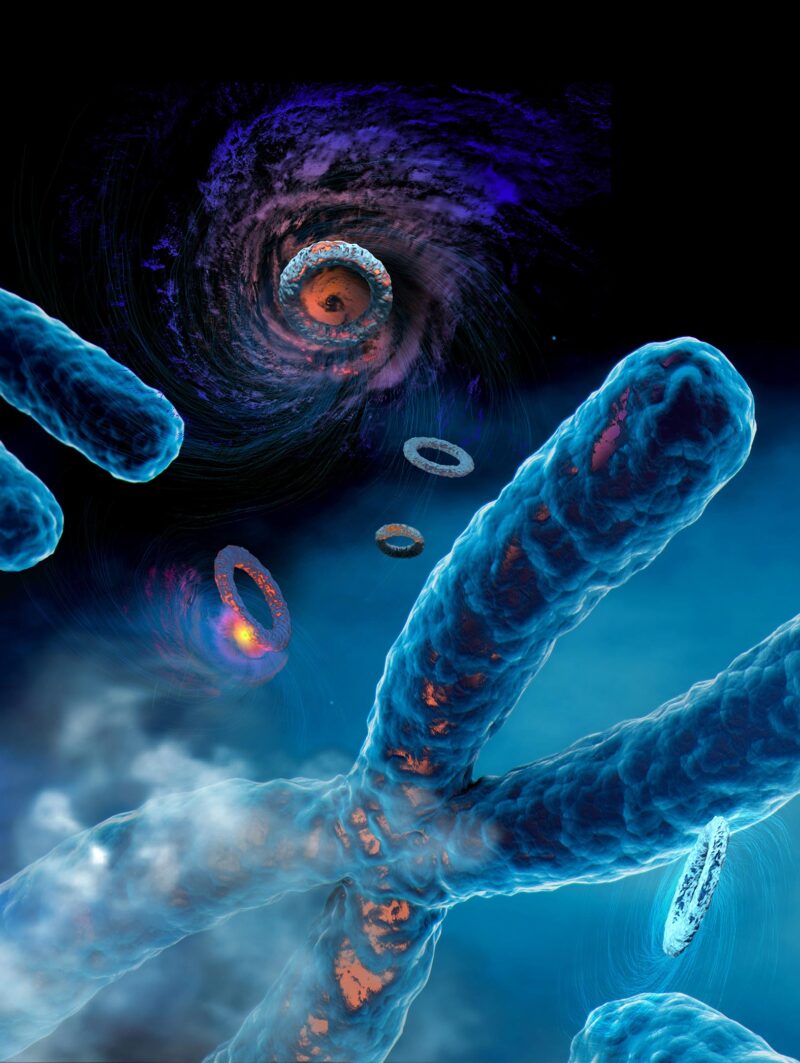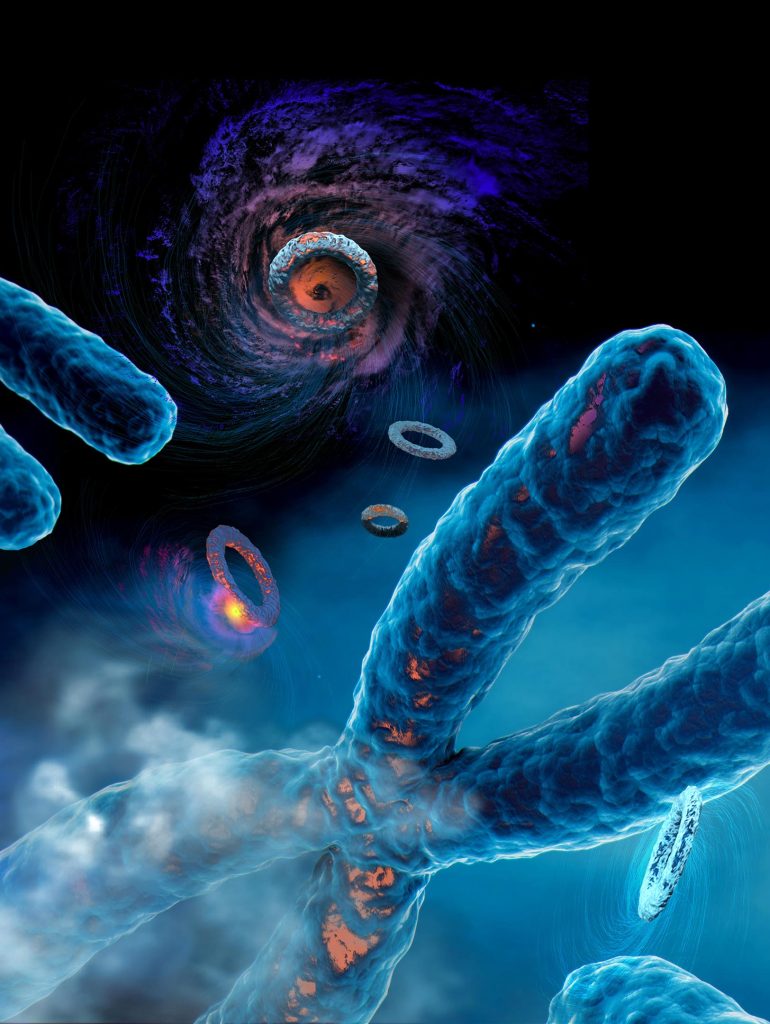
Ce rendu artistique illustre la diversité des processus mutationnels qui génèrent des mutations groupées dans le cancer humain. Sont représentés ici les kyklonas, des cyclones moléculaires qui provoquent des mutations sur l’ADN circulaire extrachromosomique (ADNe), et l’omikli, un brouillard moléculaire qui provoque des mutations sur l’ADN chromosomique linéaire. Crédit : Catherine Eng
Des chercheurs dirigés par des bio-ingénieurs de l’Université de Californie à San Diego ont identifié et caractérisé un acteur clé jusqu’alors méconnu de l’évolution du cancer : des grappes de mutations survenant dans certaines régions du génome. Les chercheurs ont découvert que ces groupes de mutations contribuent à la progression d’environ 10% des cancers humains et peuvent être utilisés pour prédire la survie des patients.
Les résultats ont été rapportés dans un article publié le 9 février 2022 dans le journal. Nature.
Les travaux mettent en lumière une classe de mutations appelées mutations somatiques groupées – groupées signifiant qu’elles se regroupent dans des zones spécifiques du génome d’une cellule, et somatiques signifiant qu’elles ne sont pas héritées, mais causées par des facteurs internes et externes tels que le vieillissement ou l’exposition aux rayons UV, par exemple.
Les mutations somatiques groupées ont jusqu’à présent été un domaine peu étudié dans le développement du cancer. Mais les chercheurs du laboratoire de Ludmil Alexandrov, professeur de bio-ingénierie et de médecine cellulaire et moléculaire à l’Université de Californie à San Diego, ont constaté que ces mutations présentaient un caractère très inhabituel qui justifiait une étude plus approfondie.
“Nous voyons généralement des mutations somatiques survenant de manière aléatoire dans le génome. Mais lorsque nous avons examiné de plus près certaines de ces mutations, nous avons constaté qu’elles se produisaient dans ces points chauds. C’est comme si on lançait des balles sur le sol et qu’on les voyait soudain se regrouper dans un seul espace”, a déclaré M. Alexandrov. “Nous ne pouvions donc pas nous empêcher de nous demander : Que se passe-t-il ici ? Pourquoi y a-t-il des points chauds ? Sont-ils cliniquement pertinents ? Nous disent-ils quelque chose sur la façon dont le cancer s’est développé ?”
“Les mutations groupées ont été largement ignorées parce qu’elles ne représentent qu’un très faible pourcentage de toutes les mutations”, a déclaré Erik Bergstrom, doctorant en bio-ingénierie dans le laboratoire d’Alexandrov et premier auteur de l’étude. “Mais en plongeant plus profondément, nous avons découvert qu’elles jouent un rôle important dans l’étiologie du cancer humain.”
Les découvertes de l’équipe ont été rendues possibles par la création de la carte la plus complète et détaillée des mutations somatiques groupées connues. Ils ont commencé par cartographier toutes les mutations (groupées et non groupées) dans les génomes de plus de 2 500 patients atteints de cancer – un effort qui, au total, a englobé 30 types de cancer différents. Les chercheurs ont créé leur carte en utilisant des approches d’intelligence artificielle de nouvelle génération développées dans le laboratoire d’Alexandrov. L’équipe a utilisé ces algorithmes pour détecter les mutations groupées chez des patients individuels et élucider les processus mutationnels sous-jacents qui donnent lieu à ces événements. Ils ont ainsi découvert que les mutations somatiques groupées contribuent à l’évolution du cancer dans environ 10 % des cancers humains.
Allant plus loin, les chercheurs ont également découvert que certains des groupes de mutations cancérigènes, en particulier ceux trouvés dans des gènes cancérigènes connus, peuvent être utilisés pour prédire la survie globale d’un patient. Par exemple, la présence de mutations groupées dans le gène BRAF – le gène pilote le plus largement observé dans le mélanome – entraîne une meilleure survie globale du patient par rapport aux individus présentant des mutations non groupées. En revanche, la présence de mutations groupées dans le gène EGFR – le gène pilote le plus largement observé dans le cancer du poumon – entraîne une diminution de la survie du patient.
“Ce qui est intéressant, c’est que nous observons une survie différentielle en fonction de la simple détection de mutations groupées dans ces gènes, et ceci est détectable avec les plateformes existantes qui sont couramment utilisées en clinique. Il s’agit donc d’un biomarqueur très simple et précis pour la survie des patients”, a déclaré Bergstrom.
“Ce travail élégant souligne l’importance de développer des approches d’IA pour élucider la biologie des tumeurs, et pour la découverte et le développement rapide de biomarqueurs en utilisant des plates-formes standard avec une traduction en ligne de mire directe vers la clinique”, a déclaré Scott Lippman, directeur du Moores Cancer Center et vice-chancelier associé pour la recherche sur le cancer et les soins à UC San Diego. “Cela souligne la force de l’UC San Diego dans la combinaison d’approches d’ingénierie en intelligence artificielle pour résoudre les problèmes actuels de la médecine du cancer.”
Un nouveau mode d’évolution du cancer
Dans cette étude, les chercheurs ont également identifié différents facteurs qui provoquent des mutations somatiques groupées. Ces facteurs comprennent les UVles radiations, la consommation d’alcool, le tabagisme et, surtout, l’activité d’un ensemble d’enzymes antivirales appelées APOBEC3.
Les enzymes APOBEC3 se trouvent généralement à l’intérieur des cellules dans le cadre de leur réponse immunitaire interne. Leur principale mission consiste à découper les virus qui pénètrent dans la cellule. Mais dans les cellules cancéreuses, les chercheurs pensent que les enzymes APOBEC3 peuvent faire plus de mal que de bien.
Les chercheurs ont découvert que les cellules cancéreuses – qui sont souvent truffées d’anneaux circulaires d’DNA (ecDNA) that harbor known cancer driver genes—have clusters of mutations occurring across individual ecDNA molecules. The researchers attribute these mutations to the activity of APOBEC3 enzymes. They hypothesize that APOBEC3 enzymes are mistaking the circular rings of ecDNA as foreign viruses and attempt to restrict and chop them up. In doing so, the APOBEC3 enzymes cause clusters of mutations to form within individual ecDNA molecules. This in turn plays a key role in accelerating cancer evolution and likely leads to drug resistance. The researchers named these rings of clustered mutations kyklonas, which is the Greek word for cyclones.
“This is a completely novel mode of oncogenesis,” said Alexandrov. Along with the team’s other findings, he explained, “this lays the foundation for new therapeutic approaches, where clinicians can consider restricting the activity of APOBEC3 enzymes and/or targeting extrachromosomal DNA for cancer treatment.”
Reference: “Mapping clustered mutations in cancer reveals APOBEC3 mutagenesis of ecDNA” by Erik N. Bergstrom, Jens Luebeck, Mia Petljak, Azhar Khandekar, Mark Barnes, Tongwu Zhang, Christopher D. Steele, Nischalan Pillay, Maria Teresa Landi, Vineet Bafna, Paul S. Mischel, Reuben S. Harris and Ludmil B. Alexandrov, 9 February 2022, Nature.
DOI: 10.1038/s41586-022-04398-6
This work was supported by a Cancer Grand Challenge award from Cancer Research UK as well as funding from the U.S. National Institutes of Health, Alfred P. Sloan Foundation, and Packard Foundation.