Par
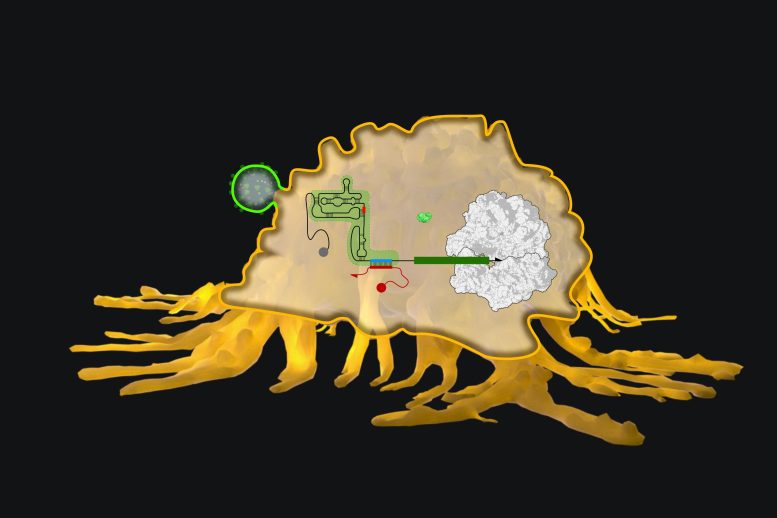
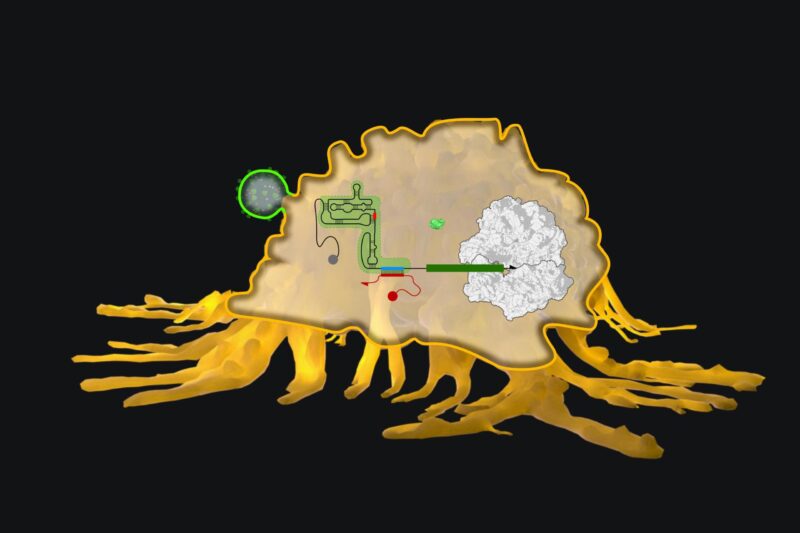
Les Toeholds eucaryotes (eToeholds) sont des éléments de contrôle conçus à base d’ARN qui, comme dans cet exemple, peuvent être spécifiquement activés par des « ARN déclencheurs » viraux pour permettre la synthèse d’une protéine rapporteur et ainsi signaler la présence du virus. À l’avenir, eToeholds pourrait être utilisé pour concevoir des thérapies à base d’ARN plus sûres et plus spécifiques, des diagnostics d’ARN et des stratégies pour enrichir les types de cellules thérapeutiques dans des approches de différenciation in vitro. Crédit : Institut Wyss de l’Université Harvard
Les eToeholds sont des éléments de contrôle conçus qui pourraient ARN thérapeutiques plus sûres, les thérapies cellulaires plus efficaces et permettent de nouvelles formes de biodétection.
Les ARN sont mieux connus comme les molécules qui traduisent les informations codées dans les gènes en protéines avec leur myriade d’activités. Cependant, en raison de leur complexité structurelle et de leur stabilité relative, l’ARN a également attiré une grande attention en tant que biomatériau précieux qui peut être utilisé pour créer de nouveaux types de thérapies, des biomarqueurs synthétiques et, bien sûr, des vaccins puissants, comme nous l’avons appris de la COVID-19[feminine pandémie.
L’introduction d’une molécule d’ARN synthétique dans une cellule lui demande essentiellement de produire une protéine souhaitée, qui peut ensuite remplir des fonctions thérapeutiques, diagnostiques et autres. Un défi clé pour les chercheurs a été de permettre uniquement aux cellules causant ou affectées par une maladie spécifique d’exprimer la protéine et pas les autres. Cette capacité pourrait considérablement rationaliser la production de la protéine dans le corps et éviter les effets secondaires indésirables.
Maintenant, une équipe de biologistes synthétiques et d’ingénieurs cellulaires dirigée par James J. Collins, Ph.D. au Wyss Institute for Biologically Inspired Engineering et au Massachusetts Institute of Technology (AVEC), a développé des eToeholds – de petits dispositifs polyvalents intégrés à l’ARN qui permettent l’expression d’une séquence codant pour une protéine liée uniquement lorsqu’un ARN spécifique à une cellule ou viral est présent. Les appareils eToehold ouvrent de multiples opportunités pour des types plus ciblés de thérapie par ARN, in vitro les approches d’ingénierie cellulaire et tissulaire et la détection de diverses menaces biologiques chez l’homme et d’autres organismes supérieurs. Les résultats sont rapportés dans Biotechnologie naturelle.
En 2014, l’équipe de Collins, avec celle de Peng Yin, Ph.D., membre du corps professoral de Wyss, a développé avec succès interrupteurs pour les bactéries qui sont exprimés à l’état désactivé et répondent à des ARN déclencheurs spécifiques en activant la synthèse d’une protéine souhaitée par la machinerie de synthèse des protéines bactériennes. Cependant, la conception bactérienne du pied ne peut pas être utilisée dans des cellules plus complexes, y compris des cellules humaines, avec leur architecture et leur appareil de synthèse de protéines plus compliqués.
« Dans cette étude, nous avons pris IRES [internal ribosome entry sites] éléments, un type d’élément de contrôle commun à certains virus, qui exploitent la machinerie de traduction des protéines eucaryotes, et les ont conçus à partir de zéro dans des dispositifs polyvalents qui peuvent être programmés pour détecter les ARN déclencheurs spécifiques aux cellules ou aux agents pathogènes chez l’homme, la levure et la plante cellules », a déclaré Collins. « eToeholds pourrait permettre des approches thérapeutiques et diagnostiques plus spécifiques et plus sûres de l’ARN non seulement chez l’homme, mais aussi chez les plantes et d’autres organismes supérieurs, et être utilisés comme outils de recherche fondamentale et de biologie synthétique. »
Les éléments de contrôle appelés « sites d’entrée internes du ribosome », en abrégé IRES, sont des séquences présentes dans l’ARN viral qui permettent aux ribosomes synthétisant les protéines de la cellule hôte d’accéder à un segment du génome viral à côté d’une séquence codant pour une protéine virale. Une fois verrouillés sur l’ARN, les ribosomes commencent à balayer la séquence codante de la protéine, tout en synthétisant simultanément la protéine en ajoutant séquentiellement les acides aminés jusqu’à sa fin de croissance.
“Nous avons fait évoluer les séquences IRES en introduisant des séquences complémentaires qui se lient les unes aux autres pour former des structures inhibitrices de paires de bases, qui empêchent le ribosome de se lier à l’IRES”, a déclaré le co-premier auteur Evan Zhao, Ph.D., qui est un Stagiaire postdoctoral dans l’équipe de Collins. « L’élément de séquence codant pour la boucle en épingle à cheveux dans eToeholds est conçu de telle sorte qu’il chevauche des séquences de capteurs spécifiques qui sont complémentaires aux ARN déclencheurs connus. Lorsque l’ARN déclencheur est présent et se lie à son complément dans les eToeholds, la boucle en épingle à cheveux s’ouvre et le ribosome peut s’activer pour faire son travail et produire la protéine.
Zhao s’est associé au co-premier auteur et membre du développement technologique de Wyss Angelo Mao, Ph.D., dans le projet eToehold, qui leur a permis de combiner leurs domaines d’expertise respectifs en biologie synthétique et en ingénierie cellulaire pour innover dans la manipulation de séquences IRES.
Au cours d’un processus d’itération rapide, ils ont pu concevoir et optimiser des eToeholds fonctionnels dans les cellules humaines et de levure, ainsi que des tests de synthèse de protéines sans cellules. Ils ont obtenu jusqu’à 16 fois l’induction de gènes rapporteurs fluorescents liés aux eToeholds exclusivement en présence de leurs ARN déclencheurs appropriés, par rapport aux ARN de contrôle.
« Nous avons conçu des eToeholds qui ont spécifiquement détecté l’infection par le virus Zika et la présence de SRAS-CoV-2 l’ARN viral dans les cellules humaines et d’autres eToeholds déclenchés par des ARN spécifiques aux cellules comme, par exemple, un ARN qui n’est exprimé que dans les mélanocytes de la peau », a déclaré Mao. « Il est important de noter que les eToeholds et les séquences codant pour les protéines souhaitées qui leur sont liées peuvent être codées de manière plus stable. ADN molécules qui, lorsqu’elles sont introduites dans les cellules, sont converties en molécules d’ARN adaptées au type d’expression protéique que nous visons. Cela étend les possibilités de livraison eToehold aux cellules cibles. »
Les chercheurs pensent que leur plate-forme eToehold pourrait aider à cibler les thérapies à ARN et certaines thérapies géniques sur des types de cellules spécifiques, ce qui est important car de nombreuses thérapies de ce type sont entravées par des toxicités excessives hors cible. De plus, cela pourrait faciliter ex vivo approches de différenciation qui guident les cellules souches le long des voies de développement pour générer des types cellulaires spécifiques pour les thérapies cellulaires et d’autres applications. La conversion de cellules souches et de cellules intermédiaires le long de nombreuses lignées cellulaires de différenciation n’est souvent pas très efficace, et les eToeholds pourraient aider à enrichir les types de cellules souhaités.
« Cette étude montre comment Jim Collins et son équipe sur la plateforme Wyss Living Cellular Device développent des outils innovants qui peuvent faire avancer le développement de thérapies cellulaires et à ARN plus spécifiques, sûres et efficaces, et ainsi avoir un impact positif sur la vie de nombreux patients, », a déclaré le directeur fondateur de Wyss, Donald Ingber, MD, Ph.D., qui est également le Judah Folkman Professeur de biologie vasculaire à la Harvard Medical School et au Boston Children’s Hospital, et professeur de bio-ingénierie à la Harvard John A. Paulson School of Engineering and Applied Sciences.
Pour en savoir plus sur cette étude, voir RNA Control Switch: Engineers Devise a Way To Selectively Turn On Gene Therapies in Human Cells.
Référence : « Éléments sensibles à l’ARN pour le contrôle de la traduction eucaryote » par Evan M. Zhao, Angelo S. Mao, Helena de Puig, Kehan Zhang, Nathaniel D. Tippens, Xiao Tan, F. Ann Ran, Isaac Han, Peter Q. Nguyen , Emma J. Chory, Tiffany Y. Hua, Pradeep Ramesh, David B. Thompson, Crystal Yuri Oh, Eric S. Zigon, Max A. English et James J. Collins, 28 octobre 2021, Biotechnologie naturelle.
DOI : 10.1038 / s41587-021-01068-2
Les autres auteurs de l’étude sont Helena de Puig, Ph.D., Kehan Zhang, Ph.D., Nathaniel Tippens, Ph.D., Xiao Tan, MD, F. Ann Ran, Ph.D., Wyss Research Assistant Isaac Han, Peter Nguyen, Ph.D., Emma Chory, Ph.D., Tiffany Hua, Pradeep Ramesh, Ph.D., Wyss Staff Scientist David Thompson, Ph.D., Crystal Yuri Oh, Eric Zigon et Max English . L’étude a été financée par des subventions de BASF, du NIH (sous la subvention # RC2 DK120535-01A1) et du Wyss Institute for Biologically Inspired Engineering.