Une équipe de scientifiques de l’UC a reconstitué l’horloge circadienne des cyanobactéries dans un tube à essai, leur permettant d’étudier les interactions moléculaires des protéines de l’horloge en temps réel et de comprendre comment ces interactions permettent à l’horloge d’exercer un contrôle sur l’expression des gènes. Crédit : Andy LiWang
L’horloge biologique reconstituée maintient des cycles quotidiens pendant des jours, permettant aux chercheurs d’étudier les interactions de ses composants.
Les cycles quotidiens dans pratiquement tous les aspects de notre physiologie sont pilotés par des horloges biologiques (également appelées horloges circadiennes) dans nos cellules. Les interactions cycliques des protéines de l’horloge maintiennent les rythmes biologiques de la vie en harmonie avec le cycle quotidien de la nuit et du jour, et cela se produit non seulement chez les humains et d’autres animaux complexes, mais même chez les organismes unicellulaires simples tels que les cyanobactéries.
Une équipe de scientifiques a maintenant reconstitué l’horloge circadienne des cyanobactéries dans un tube à essai, leur permettant d’étudier les interactions rythmiques des protéines de l’horloge en temps réel et de comprendre comment ces interactions permettent à l’horloge d’exercer un contrôle sur l’expression des gènes. Des chercheurs de trois laboratoires de l’UC Santa Cruz, de l’UC Merced et de l’UC San Diego ont collaboré à l’étude, publiée le 8 octobre 2021 dans Science.
“La reconstitution d’un processus biologique compliqué comme l’horloge circadienne à partir de zéro nous a vraiment aidés à apprendre comment les protéines de l’horloge fonctionnent ensemble et permettra une compréhension beaucoup plus approfondie des rythmes circadiens”, a déclaré Carrie Partch, professeur de chimie et de biochimie à l’UC Santa Cruz. et un auteur correspondant de l’étude.
Partch a noté que les détails moléculaires des horloges circadiennes sont remarquablement similaires des cyanobactéries aux humains. Disposer d’une horloge fonctionnelle qui peut être étudiée dans le tube à essai (« in vitro ») plutôt que dans des cellules vivantes (« in vivo ») fournit une plate-forme puissante pour explorer les mécanismes de l’horloge et comment elle réagit aux changements. L’équipe a mené des expériences sur des cellules vivantes pour confirmer que leurs résultats in vitro sont cohérents avec le fonctionnement de l’horloge dans les cyanobactéries vivantes.
« Ces résultats étaient tellement surprenants car il est courant d’avoir des résultats in vitro qui sont quelque peu incohérents avec ce qui est observé in vivo. L’intérieur des cellules vivantes est très complexe, contrairement aux conditions beaucoup plus simples in vitro », a déclaré Andy LiWang, professeur de chimie et de biochimie à l’UC Merced et auteur correspondant de l’article.
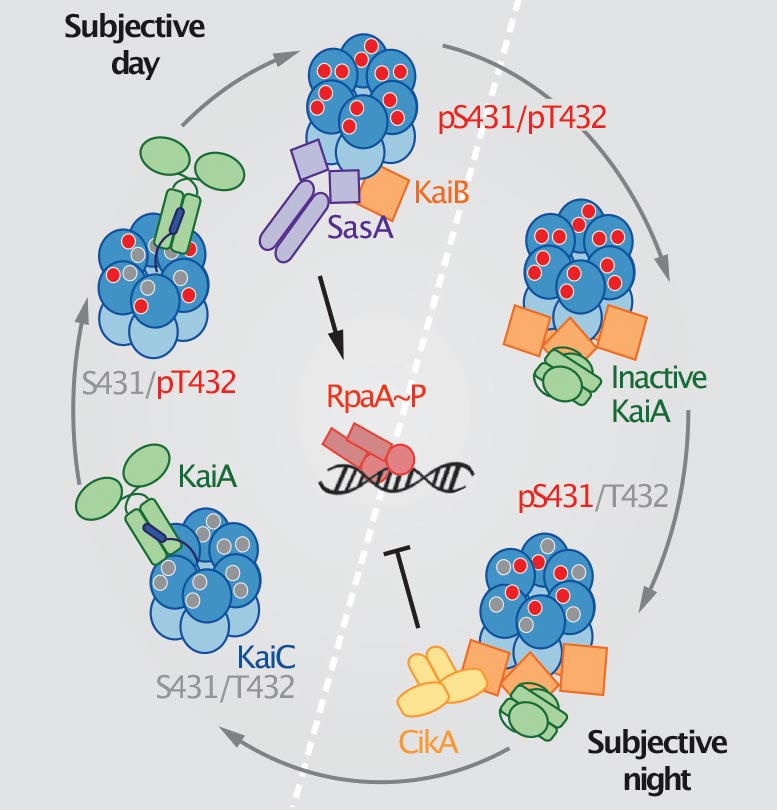
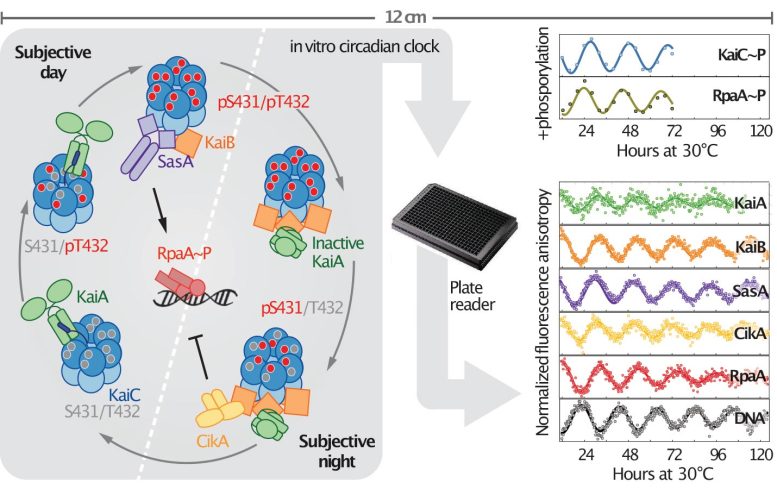
La nouvelle étude s’appuie sur les travaux antérieurs de chercheurs japonais, qui en 2005 ont reconstitué l’oscillateur circadien cyanobactérien, la boucle de base de l’horloge sur 24 heures. L’oscillateur se compose de trois protéines apparentées : KaiA, KaiB et KaiC. Dans les cellules vivantes, les signaux de l’oscillateur sont transmis par d’autres protéines pour contrôler l’expression des gènes dans un cycle circadien.
La nouvelle horloge in vitro comprend, en plus des protéines oscillatrices, deux protéines kinases (SasA et CikA), dont les activités sont modifiées en interagissant avec l’oscillateur, ainsi qu’une ADN-protéine de liaison (RpaA) et sa cible ADN.
“SasA et CikA activent et désactivent respectivement RpaA de sorte qu’il se lie et se délie rythmiquement de l’ADN”, a expliqué LiWang. « Chez les cyanobactéries, cette liaison et cette déliaison rythmiques sur plus de 100 sites différents de leur génome active et désactive l’expression de nombreux gènes importants pour la santé et la survie. »
En utilisant des techniques de marquage fluorescent, les chercheurs ont pu suivre les interactions entre tous ces composants d’horloge alors que l’ensemble du système oscille avec un rythme circadien pendant plusieurs jours, voire plusieurs semaines. Ce système a permis à l’équipe de déterminer comment SasA et CikA améliorent la robustesse de l’oscillateur, en le maintenant en marche dans des conditions dans lesquelles les protéines KaiABC cesseraient d’osciller d’elles-mêmes.
Les chercheurs ont également utilisé le système in vitro pour explorer les origines génétiques de la perturbation de l’horloge dans une souche arythmique de cyanobactéries. Ils ont identifié une seule mutation dans le gène de RpaA qui réduit l’efficacité de liaison à l’ADN de la protéine.
« Un seul aminé acide le changement du facteur de transcription fait perdre à la cellule le rythme d’expression des gènes, même si son horloge est intacte », a déclaré la co-auteure Susan Golden, directrice du Center for Circadian Biology de l’UC San Diego, dont Partch et LiWang sont également membres.
“La vraie beauté de ce projet est la façon dont l’équipe issue de trois campus UC s’est réunie pour mettre en commun des approches visant à déterminer comment une cellule peut lire l’heure”, a-t-elle ajouté. « La collaboration active s’est étendue bien au-delà des chercheurs principaux, les étudiants et les post-doctorants formés dans différentes disciplines se conférant entre eux pour partager des données génétiques, de biologie structurelle et biophysiques, s’expliquant mutuellement l’importance de leurs découvertes. La communication interdisciplinaire était aussi importante pour la réussite du projet que les compétences impressionnantes des chercheurs.
Référence : « La reconstitution d’une horloge intacte révèle des mécanismes de chronométrage circadien » 7 octobre 2021, Science.
DOI : 10.1126/science.abd4453
Les auteurs de l’article comprennent les premiers auteurs Archana Chavan et Joel Heisler à UC Merced et Jeffrey Swan à UC Santa Cruz, ainsi que les coauteurs Cigdem Sancar, Dustin Ernst et Mingxu Fang à UC San Diego, et Joseph Palacios, Rebecca Spangler, Clive Bagshaw, Sarvind Tripathi et Priya Crosby à l’UC Santa Cruz. Ce travail a été soutenu par les National Institutes of Health et la National Science Foundation.