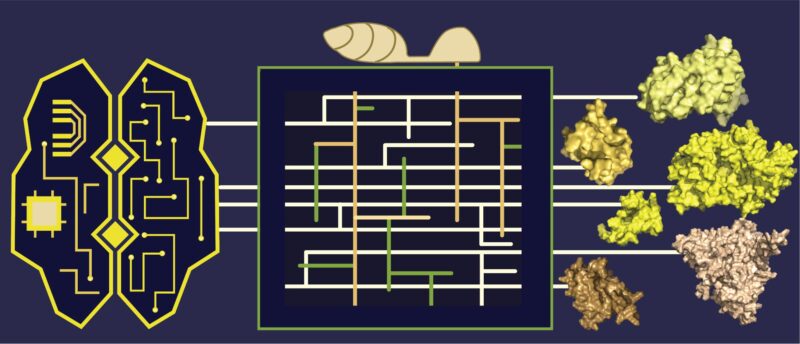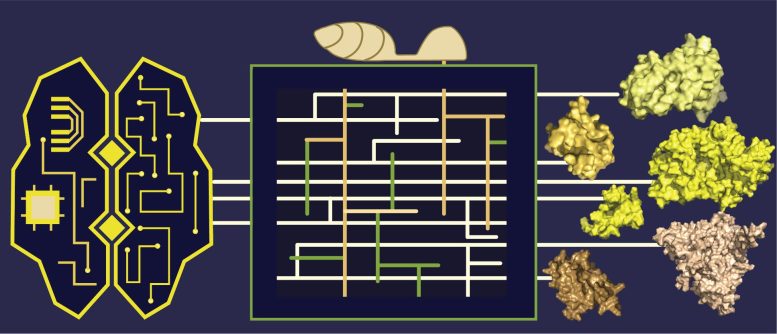
Élucidation des structures des protéines sécrétées par les phytopathogènes grâce à des outils de prédiction de structure basés sur l’apprentissage automatique. L’apprentissage automatique et l’interaction plante-pathogène ont généralement une boîte noire. Lors de la prédiction des séquences primaires d’entrée aux structures des protéines, nous ne savons pas exactement ce qui se passe. De même, nous ne comprenons pas entièrement l’interaction complexe à l’interface des plantes et des agents pathogènes. La boîte du milieu rend compte de la complexité de cette boîte noire. Crédit : Kyungyong Seong et Ksenia V. Krasileva
Des scientifiques de l’université de Californie, à Berkeley, ont récemment publié des travaux qui jettent les bases de nouvelles façons de penser l’évolution des agents pathogènes. “Notre recherche met en évidence que la modélisation sans modèle qui utilise l’apprentissage automatique est effectivement supérieure à la modélisation basée sur des modèles pour les protéines sécrétées du champignon pathogène destructeur. Magnaporthe oryzae,” a déclaré Kyungyong Seong, premier auteur de l’article publié dans l’édition du MPMI journal.
Les agents pathogènes utilisent des facteurs de virulence appelés effecteurs, qui sont importants pour la survie de l’agent pathogène. La modélisation homologique est l’une des méthodes les plus utilisées, mais elle nécessite l’utilisation de modèles de structures d’effecteurs résolus et la résolution de toutes les structures d’effecteurs est une tâche trop ardue. Il existe trop de protéines effectrices codées dans les génomes des agents pathogènes pour que l’on puisse se contenter de résoudre expérimentalement chacune de ces structures.
Seong et sa collègue Ksenia V. Krasileva ont utilisé une nouvelle méthode de prédiction de structure qui a permis de modéliser 500 protéines sécrétées qui n’avaient pas été prédites par la méthode basée sur les modèles.
“Environ 70% des 1 854 protéines sécrétées ont été modélisées dans notre étude, et leurs structures fournissent une couche supplémentaire d’informations sur les effecteurs en fonction de leur similarité entre elles ou avec d’autres structures de protéines résolues”, a déclaré Krasileva. “Nous démontrons que les nouvelles méthodes de prédiction de structure s’appliquent bien au problème du déchiffrage des facteurs de virulence des agents pathogènes et d’autres protéines sécrétées qui présentent souvent peu de similarité de séquence entre elles ou avec d’autres protéines.”
Cette nouvelle méthode permet aux scientifiques de cartographier des milliers de protéines sécrétées et d’établir les liens évolutifs manquants entre elles. “Nous pensons que notre recherche a été la première à appliquer le concept de génomique structurelle sur un agent pathogène végétal dans la nouvelle ère de la prédiction de structure par apprentissage automatique”, a déclaré Seong.
“Au fur et à mesure que la précision de la prédiction de structure s’améliore, il deviendra plus courant de voir des articles qui intègrent des données de prédiction de structure de protéines à grande échelle”, a prédit Krasileva. “Notre article peut susciter des idées sur la façon d’utiliser ces données, ce qui conduira certains scientifiques à explorer des opportunités avant les autres.”
Les chercheurs ont également découvert qu’il existe de nombreux effecteurs structurellement similaires, sans lien avec la séquence, dans les cellules de l’ADN. M. oryzaeet des effecteurs structurellement similaires sont trouvés dans d’autres phytopathogènes. Cela suggère que les agents pathogènes peuvent s’appuyer sur un ensemble d’effecteurs d’origine commune mais dont les séquences ont largement divergé au cours de l’évolution pour infecter les plantes.
Référence : Computational Structural Genomics Unravels Common Folds and Novel Families in the Secretome of Fungal Phytopathogen” (La génomique structurelle computationnelle révèle des plis communs et de nouvelles familles dans le sécrétome des phytopathogènes fongiques). Magnaporthe oryzae“par Kyungyong Seong et Ksenia V. Krasileva, 10 novembre 2021, MPMI journal.
DOI : 10.1094/MPMI-03-21-0071-R