Deux nouvelles méthodes permettent l’édition CRISPR de gènes dans plusieurs types de cellules simultanément.
À ce jour, les enzymes CRISPR ont été utilisées pour modifier les génomes d’un type de cellule à la fois : elles coupent, suppriment ou ajoutent des gènes à un type spécifique de cellule dans un tissu ou un organe, par exemple, ou à un type de microbe en croissance dans un tube à essai.
Maintenant le Université de Californie, Berkeley, le groupe qui a inventé la technologie d’édition du génome CRISPR-Cas9 il y a près de 10 ans a trouvé un moyen d’ajouter ou de modifier des gènes au sein d’une communauté de nombreuses espèces différentes simultanément, ouvrant la porte à ce que l’on pourrait appeler « l’édition communautaire ».
Bien que cette technologie soit encore exclusivement appliquée en laboratoire, elle pourrait être utilisée à la fois pour modifier et pour suivre les microbes modifiés au sein d’une communauté naturelle, comme dans l’intestin ou sur les racines d’une plante où se rassemblent des centaines ou des milliers de microbes différents. Un tel suivi devient nécessaire alors que les scientifiques parlent de modifier génétiquement les populations microbiennes : insérer des gènes dans des microbes dans l’intestin pour résoudre les problèmes digestifs, par exemple, ou modifier l’environnement microbien des cultures pour les rendre plus résistantes aux ravageurs.
Sans moyen de suivre les insertions de gènes – en utilisant un code-barres, dans ce cas – ces gènes insérés pourraient se retrouver n’importe où, car les microbes partagent régulièrement des gènes entre eux.
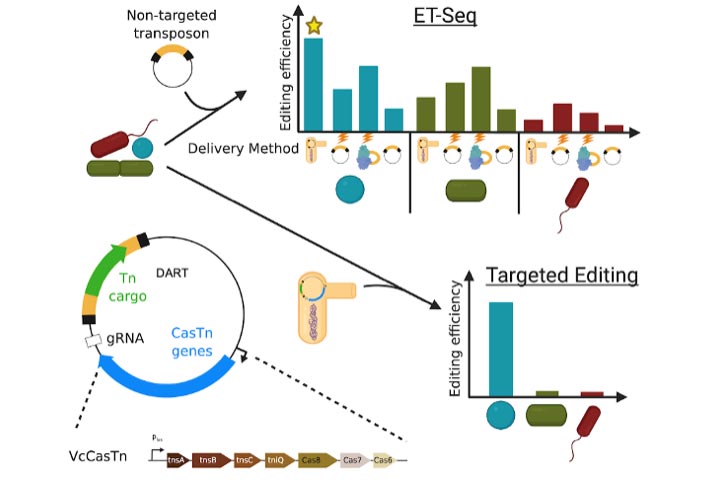
Pour éditer avec succès des gènes au sein de plusieurs membres d’une communauté microbienne, les scientifiques de l’UC Berkeley ont dû développer deux nouvelles méthodes : le séquençage de la transformation environnementale (ET-Seq), en haut, qui leur a permis d’évaluer l’éditabilité de microbes spécifiques ; et la transposase CRISPR-Cas (DART) tout-en-un d’édition d’ADN, qui a permis une insertion d’ADN ciblée hautement spécifique dans un emplacement du génome défini par un ARN guide. Le système DART est doté d’un code-barres et compatible avec ET-Seq, de sorte que, lorsqu’ils sont utilisés ensemble, les scientifiques peuvent insérer, suivre et évaluer l’efficacité et la spécificité de l’insertion. Crédit : laboratoire Jill Banfield, UC Berkeley
« Casser et changer ADN au sein de micro-organismes isolés a été essentiel pour comprendre ce que fait cet ADN », a déclaré Benjamin Rubin, chercheur postdoctoral à l’UC Berkeley. «Ce travail contribue à apporter cette approche fondamentale aux communautés microbiennes, qui sont beaucoup plus représentatives de la façon dont ces microbes vivent et fonctionnent dans la nature.»
Alors que la possibilité de modifier de nombreux types de cellules ou de microbes à la fois pourrait être utile dans les systèmes actuels à l’échelle de l’industrie – les bioréacteurs pour la culture de cellules en vrac, par exemple, l’application la plus immédiate peut être un outil pour comprendre la structure de des communautés complexes de bactéries, d’archées et de champignons, et le flux de gènes au sein de ces diverses populations.
« À terme, nous pourrons peut-être éliminer les gènes qui causent des maladies dans vos bactéries intestinales ou rendre les plantes plus efficaces en créant leurs partenaires microbiens », a déclaré le boursier postdoctoral Brady Cress. “Mais probablement, avant de faire cela, cette approche nous donnera une meilleure compréhension du fonctionnement des microbes au sein d’une communauté.”
Rubin et Cress – tous deux dans le laboratoire de l’inventeur CRISPR-Cas9 Jennifer Doudna – et Spencer Diamond, un scientifique du projet à l’Innovative Genomics Institute (IGI), sont les co-premiers auteurs d’un article décrivant la technique parue aujourd’hui (6 décembre). ) dans la revue Microbiologie naturelle.
Du recensement à l’édition
Diamond travaille dans le laboratoire de Jill Banfield, une géomicrobiologiste pionnière dans le domaine du séquençage communautaire, ou métagénomique : séquençage au fusil de chasse de tout l’ADN d’une communauté complexe de microbes et assemblage de cet ADN dans les génomes complets de tous ces organismes, dont certains probablement n’ont jamais été vus auparavant et dont beaucoup sont impossibles à cultiver dans un plat de laboratoire.
Le séquençage métagénomique a énormément progressé au cours des 15 dernières années. En 2019, Diamond a rassemblé 10 000 génomes individuels de près de 800 espèces microbiennes à partir d’échantillons de sol prélevés dans une prairie de prairie du nord de la Californie.
Mais il compare cela à un recensement de la population : il fournit des informations sans précédent sur les microbes présents dans quelles proportions et les fonctions que ces microbes pourraient remplir au sein de la communauté. Et cela vous permet de déduire des interactions compliquées entre les organismes et comment ils peuvent travailler ensemble pour obtenir des avantages importants pour l’écosystème, tels que la fixation de l’azote. Mais ces observations ne sont que des hypothèses ; de nouvelles méthodes sont nécessaires pour tester réellement ces fonctions et interactions au niveau communautaire, a déclaré Diamond.
“Il y a cette idée de transferts métaboliques – qu’aucun microbe individuel n’exécute une énorme chaîne de fonctions métaboliques, mais pour la plupart, chaque organisme individuel effectue une seule étape d’un processus, et qu’il doit y avoir un certain transfert de métabolites entre les organismes », a-t-il déclaré. « C’est l’hypothèse, mais comment le prouver réellement ? Comment pouvons-nous en arriver à un point où nous ne nous contentons plus de regarder les oiseaux, nous pouvons en fait faire quelques manipulations et voir ce qui se passe ? Ce fut la genèse du montage communautaire.
L’équipe de recherche était dirigée par Banfield, professeur de sciences de la Terre et des planètes à l’UC Berkeley et de sciences, politiques et gestion de l’environnement, et Jennifer Doudna, professeure de biologie moléculaire et cellulaire et de chimie à l’UC Berkeley, chercheur et co-lauréat du Howard Hughes Medical Institute. du prix Nobel de chimie 2020 pour l’invention CRISPR-Cas9 édition du génome.
L’équipe a d’abord développé une approche pour déterminer quels microbes dans une communauté sont réellement sensibles à l’édition de gènes. La technique de criblage développée par Rubin et Diamond, appelée ET-seq (séquençage de transformation environnementale), utilise comme sonde un transposon, ou gène sauteur, qui s’insère facilement de manière aléatoire dans de nombreux génomes microbiens. En séquençant l’ADN de la communauté avant et après l’introduction du transposon, ils ont pu identifier les espèces de microbes capables d’incorporer le gène du transposon. L’approche était basée sur des techniques développées par le co-auteur Adam Deutschbauer au Lawrence Berkeley National Laboratory. Dans une expérience impliquant une communauté de neuf microbes différents, ils ont réussi à insérer le même transposon dans cinq d’entre eux en utilisant différentes méthodes de transformation.
Cress a ensuite développé un système de livraison ciblé appelé DNA-editing All-in-one ARN-Guided CRISPR Cas Transposase (DART) qui utilise une enzyme CRISPR-Cas similaire à CRISPR-Cas9 pour se concentrer sur une séquence d’ADN spécifique et insérer un transposon à code-barres.
Pour tester la technique DART avec une communauté microbienne plus réaliste, les chercheurs ont prélevé un échantillon de selles d’un nourrisson et l’ont cultivé pour créer une communauté stable composée principalement de 14 types différents de micro-organismes. Ils ont pu éditer individuellement E. coli souches au sein de cette communauté, ciblant les gènes qui ont été associés à la maladie.
Les chercheurs espèrent utiliser cette technique pour comprendre des communautés artificielles et simples, telles qu’une plante et son microbiome associé, dans une boîte fermée. Ils peuvent ensuite manipuler les gènes de la communauté au sein de ce système fermé et suivre l’effet sur leurs microbes à code-barres. Ces expériences sont un aspect d’un programme de 10 ans financé par le ministère de l’Énergie appelé m-CAFEs, pour Microbial Community Analysis and Functional Evaluation in Soils, qui cherche à comprendre la réponse d’un microbiome d’herbe simple aux changements externes. Banfield, Doudna et Deutschbauer font partie du projet m-CAFEs.
Référence : « Edition du génome spécifique à l’espèce et au site dans les communautés bactériennes complexes » par Benjamin E. Rubin, Spencer Diamond, Brady F. Cress, Alexander Crits-Christoph, Yue Clare Lou, Adair L. Borges, Haridha Shivram, Christine He, Michael Xu, Zeyi Zhou, Sara J. Smith, Rachel Rovinsky, Dylan CJ Smock, Kimberly Tang, Trenton K. Owens, Netravathi Krishnappa, Rohan Sachdeva, Rodolphe Barrangou, Adam M. Deutschbauer, Jillian F. Banfield et Jennifer A. Doudna, 6 décembre 2021, Microbiologie naturelle.
DOI : 10.1038/s41564-021-01014-7
La recherche a été soutenue par m-CAFEs (DE-AC02-05CH11231) et le National Institute of General Medical Sciences des National Institutes of Health (F32GM134694, F32GM131654).
Les autres co-auteurs de l’article sont Alexander Crits-Christoph, Yue Clare Lou, Adair Borges, Haridha Shivram, Christine He, Michael Xu, Zeyi Zhou, Sara Smith, Rachel Rovinsky, Dylan Smock, Kimberly Tang, Netravathi Krishnappa et Rohan Sachdeva de UC Berkeley; Trenton Owens du laboratoire de Berkeley ; et Rodolphe Barrangou de Université d’État de Caroline du Nord.




