Une nouvelle étude menée par des chercheurs de Penn State a révélé les étapes chimiques impliquées dans l’ajout d’un marqueur important – un groupe méthyle de soufre – pour transférer l’ARN, un processus qui, s’il est perturbé chez l’homme, peut entraîner des maladies neuronales, du diabète et des cancers. Crédit : Booker Lab, Penn State
Les étapes chimiques d’un important processus de modification cellulaire qui ajoute une étiquette chimique à certains ARN ont été révélées dans une nouvelle étude. Interférer avec ce processus chez l’homme peut entraîner des maladies neuronales, du diabète et des cancers. Une équipe de recherche, dirigée par des chimistes de Penn State, a imagé une protéine qui facilite cette ARN modification chez les bactéries, permettant aux chercheurs de reconstituer la méthode. Un article décrivant le processus de modification a été publié le 15 septembre 2021 dans la revue La nature .
Les ARN de transfert (ARNt) seraient les ARN qui «liraient» le code génétique et le traduiraient en une séquence de protéines pour produire une protéine. L’ajout d’une étiquette chimique (un groupe méthyle de soufre) à un emplacement particulier sur certains ARNt améliore leur capacité à traduire l’ARN messager en protéines. Lorsque ce processus de modification, appelé méthylthiolation, ne se produit pas correctement, des erreurs peuvent être incorporées dans les protéines résultantes, ce qui, chez l’homme, peut entraîner une infection neuronale, un cancer et un risque accru de développer un diabète de type 2.
“La méthylthiolation est omniprésente dans les bactéries, les plantes et les animaux”, a déclaré Squire Booker, biochimiste à Penn State et chercheur au Howard Hughes Medical Institute qui a utilisé la diode électroluminescente pour l’équipe de recherche. « Dans cette étude, nous avons déterminé la structure d’une protéine appelée MiaB pour mieux comprendre son rôle dans la facilitation de cet important processus de modification chez les bactéries. ”
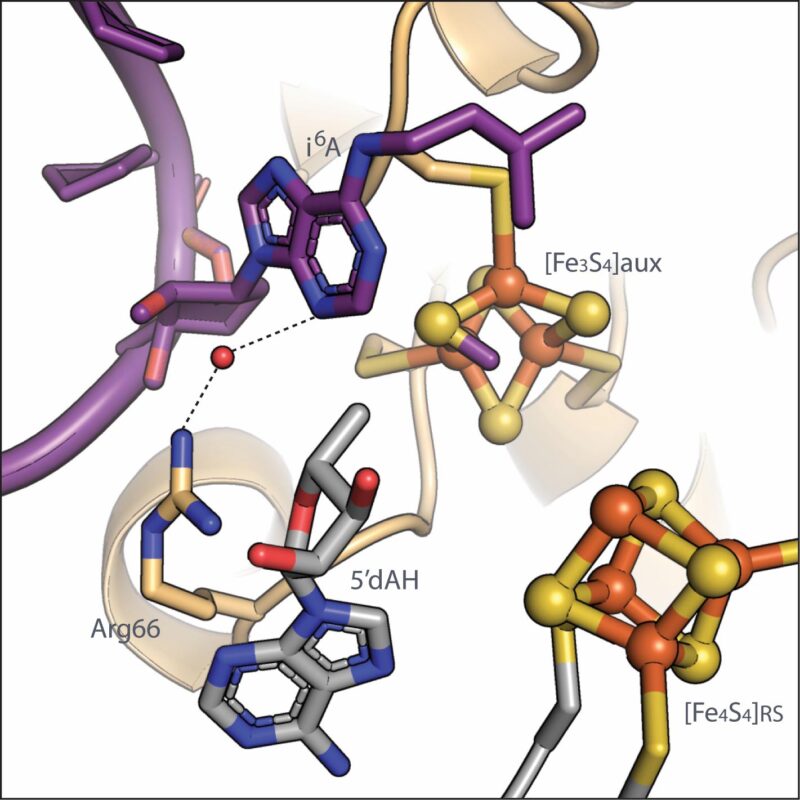
La protéine MiaB de la bactérie Bacteroides uniforms fait partie de la famille des enzymes radicalaires SAM (S-adénosylméthionine). Les enzymes SAM radicales utilisent en moyenne l’un de leurs propres clusters fer-soufre pour convertir une molécule SAM en un «radical libre» qui aide à faire avancer la réaction. Contrairement à presque toutes les autres enzymes SAM radicales, MiaB contient deux clusters fer-soufre : un cluster SAM radical et un cluster auxiliaire, où se déroule une grande partie de la chimie complexe.
L’imagerie de MiaB en action avec des molécules SAM et de l’ARNt à plusieurs moments au cours de la méthylthiolation a permis aux chercheurs de déduire les étapes chimiques au cours du processus de modification. Premièrement, une molécule de SAM fait don de son groupe méthyle au cluster auxiliaire fer-soufre sur MiaB.
« La source du soufre atome attaché à l’ARNt a été controversé, mais nos structures révèlent qu’un groupe méthyle de SAM se fixe à un atome de soufre sur le cluster auxiliaire fer-soufre de MiaB », a déclaré Olga Esakova, professeure adjointe de recherche en chimie à Penn State et premier auteur de l’article . «Ce groupe méthyle et le soufre auquel il se fixe sur MiaB sont en fin de compte ce qui est transféré à l’ARNt, mais certaines étapes supplémentaires se produisent avant que l’ARNt puisse accepter le groupe méthylthio. ”
L’ajout d’un électron fragmente une deuxième molécule de SAM en un radical libre. Le radical nécessite finalement un atome d’hydrogène de l’ARNt, qui sera remplacé par le groupe méthylthio sur MiaB.
« Initialement, l’hydrogène sur l’ARNt n’est pas positionné de manière à permettre à la fois l’accès au radical qui l’élimine et l’utilisation du groupe méthylthio qui doit être transféré, car l’hydrogène et les atomes attachés à proximité sont tous alignés dans le même avion », a déclaré Booker. “Nos structures montrent que le groupe méthylthio sur le cluster auxiliaire de MiaB induit une altération de la géométrie à cet endroit de l’ARNt subissant une méthylthiolation, qui se transforme en une forme plus tétraédrique, l’hydrogène étant dans une position optimale pour être arraché par le radical et le groupe méthylthio dans une position optimale pour un transfert ultérieur. ”
Le résultat de ces étapes est l’ARNt avec le groupe méthylthio ajouté et une modification réussie.
Ensuite, les chercheurs espèrent identifier comment un cluster auxiliaire est reconstruit après chaque rotation afin que le processus puisse se dérouler sur plusieurs tours. Ils étudient également des protéines analogues qui jouent un rôle similaire dans le processus de modification chez l’homme.
Référence : « Base structurelle de la méthylthiolation de l’ARNt par l’enzyme radicale SAM MiaB » par Olga A. Esakova, Tyler L. Grove, Neela H. Yennawar, Arthur J. Arcinas, Bo Wang, Carsten Krebs, Steven C. Almo et Squire J. Booker, 15 septembre 2021, La nature .
DOI : 10. 1038 / s41586-021-03904-6
Booker est professeur de chimie et de biochimie et biologie moléculaire à l’Université Evan Pugh et titulaire de la chaire Eberly Distinguished Family in Science à Penn State. En plus de Booker et Esakova, l’équipe de recherche de Penn State comprend Neela Yennawar, directrice des installations centrales de cristallographie aux rayons X et de calorimétrie biologique automatisée de Penn State ; Arthur Arcinas, étudiant diplômé au moment de la recherche ; Bo Wang, professeur adjoint de recherche en chimie ; et Carsten Krebs, professeur de chimie et de biochimie et biologie moléculaire. L’équipe comprend également Tyler Grove et Steven Almo de l’Albert Einstein College of Medicine.
Cette recherche a été soutenue par le Howard Hughes Medical Institute, les National Institutes of Health, la National Science Foundation, le Penn State Eberly College of Science, la Price Family Foundation et les Penn State Huck Institutes of the Life Sciences.