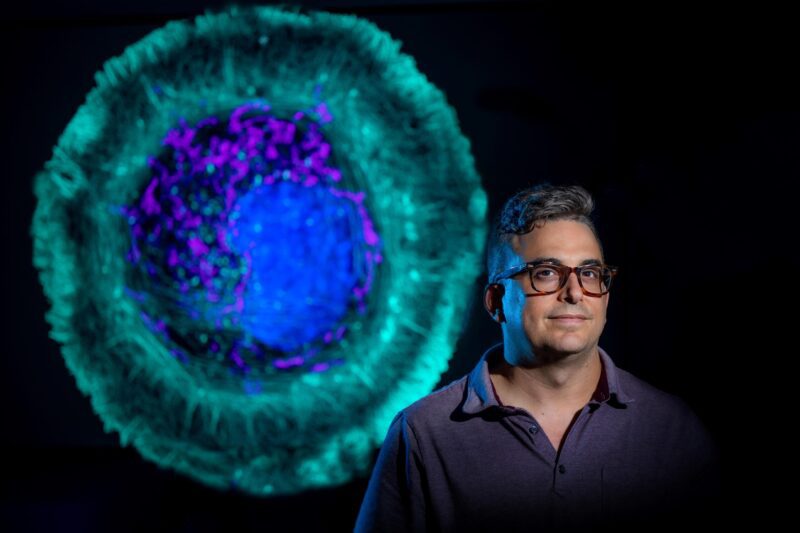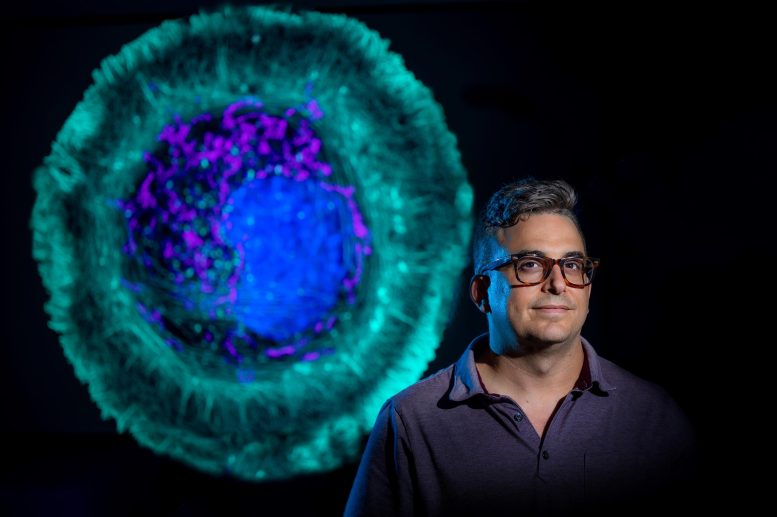
Dr Eric A. Vitriol. Crédit : Michael Holahan, Université Augusta
Un nouveau « pipeline d’analyse d’images » donne aux scientifiques un nouvel aperçu rapide de la façon dont la maladie ou les blessures ont modifié le corps, jusqu’à la cellule individuelle.
Il s’appelle TDAExplore, qui utilise l’imagerie détaillée fournie par la microscopie, l’associe à un domaine chaud des mathématiques appelé topologie, qui donne un aperçu de la façon dont les choses sont organisées et de la puissance analytique de l’intelligence artificielle pour donner, par exemple, une nouvelle perspective sur changements dans une cellule résultant de la SLA et où dans la cellule ils se produisent, explique le Dr Eric Vitriol, biologiste cellulaire et neuroscientifique au Medical College of Georgia.
Il s’agit d’une “option accessible et puissante” pour utiliser un ordinateur personnel pour générer des informations quantitatives – mesurables et par conséquent objectives – à partir d’images microscopiques qui pourraient probablement être également appliquées à d’autres techniques d’imagerie standard telles que les rayons X et les tomodensitogrammes, rapportent-ils dans le journal Motifs.
“Nous pensons que c’est un progrès passionnant dans l’utilisation des ordinateurs pour nous donner de nouvelles informations sur la façon dont les ensembles d’images sont différents les uns des autres”, a déclaré Vitriol. “Quels sont les changements biologiques réels qui se produisent, y compris ceux que je ne pourrai peut-être pas voir, parce qu’ils sont trop infimes, ou parce que j’ai une sorte de préjugé sur l’endroit où je devrais regarder.”
Au moins dans le département d’analyse des données, les ordinateurs font battre notre cerveau, dit le neuroscientifique, non seulement dans leur objectivité mais dans la quantité de données qu’ils peuvent évaluer. La vision par ordinateur, qui permet aux ordinateurs d’extraire des informations d’images numériques, est un type d’apprentissage automatique qui existe depuis des décennies, c’est pourquoi lui et son collègue et collègue auteur correspondant, le Dr Peter Bubenik, mathématicien au Université de Floride et un expert en analyse de données topologiques, a décidé d’associer le détail de la microscopie à la science de la topologie et à la puissance analytique de l’IA. La topologie et Bubenik étaient essentiels, dit Vitriol.
La topologie est «parfaite» pour l’analyse d’images, car les images sont constituées de motifs, d’objets disposés dans l’espace, dit-il, et l’analyse de données topologiques (la TDA dans TDAExplore) aide également l’ordinateur à reconnaître la configuration du terrain, dans ce cas où l’actine – une protéine et un élément constitutif essentiel des fibres, ou filaments, qui aident à donner forme et mouvement aux cellules – a bougé ou changé de densité. C’est un système efficace qui, au lieu de prendre littéralement des centaines d’images pour apprendre à l’ordinateur à les reconnaître et à les classer, peut apprendre sur 20 à 25 images.
Une partie de la magie est que l’ordinateur apprend maintenant les images en morceaux qu’ils appellent des patchs. La décomposition des images de microscopie en ces morceaux permet une classification plus précise, moins de formation de l’ordinateur sur ce à quoi ressemble «normal» et, finalement, l’extraction de données significatives, écrivent-ils.
Il ne fait aucun doute que la microscopie, qui permet d’examiner de près des choses invisibles à l’œil humain, produit de belles images détaillées et des vidéos dynamiques qui sont un pilier pour de nombreux scientifiques. « Vous ne pouvez pas avoir une faculté de médecine sans installations de microscopie sophistiquées », dit-il.
Mais pour comprendre d’abord ce qui est normal et ce qui se passe dans les états pathologiques, Vitriol a besoin d’une analyse détaillée des images, comme le nombre de filaments ; où se trouvent les filaments dans les cellules – près du bord, du centre, dispersés partout – et si certaines régions cellulaires en ont plus.
Les schémas qui émergent dans ce cas lui indiquent où se trouve l’actine et comment elle est organisée – un facteur majeur de sa fonction – et où, comment et si elle a changé avec la maladie ou les dommages.
Alors qu’il regarde le regroupement d’actine autour des bords d’une cellule du système nerveux central, par exemple, l’assemblage lui dit que la cellule s’étend, se déplace et envoie des projections qui deviennent son bord d’attaque. Dans ce cas, la cellule, qui a dormi pour l’essentiel dans un plat, peut s’étaler et se dégourdir les pattes.
Certains des problèmes rencontrés par les scientifiques analysant directement les images et calculant ce qu’ils voient incluent le fait que cela prend du temps et que même les scientifiques ont des biais.
À titre d’exemple, et en particulier avec tant d’action qui se passe, leurs yeux peuvent se poser sur le familier, dans le cas de Vitriol, cette actine au bord d’attaque d’une cellule. Alors qu’il regarde à nouveau le cadre sombre autour de la périphérie de la cellule indiquant clairement l’actine qui s’y concentre, cela pourrait impliquer qu’il s’agit du principal point d’action.
« Comment puis-je savoir que lorsque je décide de ce qui est différent, c’est la chose la plus différente ou est-ce juste ce que je voulais voir ? » il dit. “Nous voulons y apporter une objectivité informatique et nous voulons apporter un degré plus élevé de reconnaissance de formes dans l’analyse des images.”
L’IA est connue pour être capable de « classifier » des choses, comme reconnaître un chien ou un chat à chaque fois, même si l’image est floue, en apprenant d’abord plusieurs millions de variables associées à chaque animal jusqu’à ce qu’il connaisse un chien lorsqu’il en voit un, mais il ne peut pas dire pourquoi c’est un chien. Cette approche, qui nécessite tant d’images à des fins d’entraînement et ne fournit toujours pas beaucoup de statistiques d’images, ne fonctionne pas vraiment pour ses objectifs, c’est pourquoi lui et ses collègues ont créé un nouveau classificateur limité à l’analyse de données topologiques.
L’essentiel est que le couplage unique utilisé dans TDAExplore indique de manière efficace et objective aux scientifiques où et dans quelle mesure l’image de la cellule perturbée diffère de l’image d’entraînement, ou de l’image normale, des informations qui fournissent également de nouvelles idées et directions de recherche, dit-il.
De retour à l’image de la cellule qui montre le regroupement d’actine le long de son périmètre, alors que le « bord d’attaque » était clairement différent avec les perturbations, TDAExplore a montré que certains des changements les plus importants se trouvaient en fait à l’intérieur de la cellule.
“Une grande partie de mon travail consiste à essayer de trouver des motifs dans des images difficiles à voir”, explique Vitriol, “Parce que je dois identifier ces motifs afin de pouvoir trouver un moyen d’obtenir des chiffres à partir de ces images.” Ses résultats consistent à déterminer comment fonctionne le cytosquelette d’actine, pour lequel les filaments fournissent l’échafaudage et qui à son tour fournit un soutien aux neurones, et ce qui ne va pas dans des conditions comme la SLA.
Certains de ces modèles d’apprentissage automatique qui nécessitent des centaines d’images pour former et classer les images ne décrivent pas quelle partie de l’image a contribué à la classification, écrivent les enquêteurs. Ces énormes quantités de données qui doivent être analysées et qui peuvent inclure environ 20 millions de variables, nécessitent un super ordinateur. Le nouveau système a plutôt besoin de relativement peu d’images à haute résolution et caractérise les « patchs » qui ont conduit à la classification sélectionnée. En quelques minutes, l’ordinateur personnel standard du scientifique peut compléter le nouveau pipeline d’analyse d’images.
L’approche unique utilisée dans TDAExplore indique objectivement aux scientifiques où et dans quelle mesure l’image perturbée diffère de l’image d’entraînement, des informations qui fournissent également de nouvelles idées et directions de recherche, dit-il.
La capacité d’obtenir des informations plus nombreuses et de meilleure qualité à partir des images signifie en fin de compte que les informations générées par des scientifiques fondamentaux comme Vitriol, qui changent souvent en fin de compte ce qui est considéré comme les faits d’une maladie et la façon dont elle est traitée, sont plus précises. Cela pourrait inclure la capacité de reconnaître les changements, comme ceux que le nouveau système a signalés à l’intérieur de la cellule, qui ont été précédemment négligés.
Actuellement, les scientifiques appliquent des taches pour permettre un meilleur contraste, puis utilisent un logiciel pour extraire des informations sur ce qu’ils voient dans les images, comme la façon dont l’actine est organisée en une structure plus grande, dit-il.
« Nous avons dû trouver une nouvelle façon d’obtenir des données pertinentes à partir d’images et c’est le sujet de cet article. »
Référence : « TDAExplore : Quantitative analysis of fluorescence microscopy images through topology-based machine learning » par Parker Edwards, Kristen Skruber, Nikola Milicevic, James B. Heidings, Tracy-Ann Read, Peter Bubenik et Eric A. Vitriol, 12 octobre 2021, Motifs.
DOI: 10.1016/j.patter.2021.100367
L’étude publiée fournit tous les éléments permettant aux autres scientifiques d’utiliser TDAExplore.
La recherche a été soutenue par les National Institutes of Health.