Soumya Kannan est une étudiante diplômée du Centre Yang-Tan 2021-2022 dans le laboratoire du professeur Feng Zhang du MIT et co-premier auteur avec Han Altae-Tran d’une étude rapportant une nouvelle classe de systèmes de modification de l’ADN programmables connus sous le nom d’OMEGA. . Crédit : Caitlin Cunningham
Nouvelles protéines d’édition de gènes programmables trouvées en dehors des systèmes CRISPR
Les chercheurs trouvent ARN-les enzymes guidées sont plus diverses et plus répandues qu’on ne le croyait auparavant.
Au cours de la dernière décennie, les scientifiques ont adapté les systèmes CRISPR des microbes à la technologie d’édition de gènes, un système précis et programmable pour modifier ADN. Désormais, les scientifiques de AVECLe McGovern Institute for Brain Research et le Broad Institute du MIT et de Harvard ont découvert une nouvelle classe de systèmes de modification d’ADN programmables appelés OMEGA (Obligate Mobile Element Guided Activity), qui peuvent naturellement être impliqués dans le brassage de petits morceaux d’ADN dans les génomes bactériens.
Ces anciennes enzymes coupant l’ADN sont guidées vers leurs cibles par de petits morceaux d’ARN. Bien qu’ils proviennent de bactéries, ils ont maintenant été conçus pour fonctionner dans des cellules humaines, ce qui suggère qu’ils pourraient être utiles dans le développement de thérapies d’édition de gènes, d’autant plus qu’ils sont petits (environ 30 % de la taille de Cas9), ce qui les rend plus faciles à utiliser. livrer aux cellules que les enzymes plus volumineuses. La découverte, rapportée le 9 septembre 2021, dans la revue Science, fournit la preuve que les enzymes naturelles guidées par l’ARN sont parmi les protéines les plus abondantes sur Terre, pointant vers un vaste nouveau domaine de la biologie qui est prêt à conduire la prochaine révolution dans la technologie d’édition du génome.
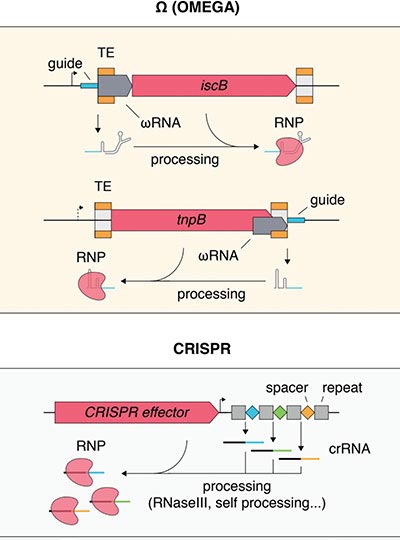
Comparaison des systèmes (OMEGA) avec d’autres systèmes connus guidés par l’ARN. Contrairement aux systèmes CRISPR, qui capturent les séquences d’espacement et les stockent dans le locus de la puce CRISPR, les systèmes peuvent transposer leurs loci (ou loci agissant en trans) en séquences cibles, convertissant les cibles en guides RNA. Crédit : Avec l’aimable autorisation des chercheurs
La recherche a été dirigée par Feng Zhang, chercheur de McGovern, professeur de neurosciences James et Patricia Poitras au MIT, chercheur au Howard Hughes Medical Institute et membre du Core Institute du Broad Institute. L’équipe de Zhang explore la diversité naturelle à la recherche de nouveaux systèmes moléculaires pouvant être programmés de manière rationnelle.
“Nous sommes très excités par la découverte de ces enzymes programmables répandues, qui se sont toujours cachées sous notre nez”, a déclaré Zhang. “Ces résultats suggèrent la possibilité alléchante qu’il existe de nombreux autres systèmes programmables qui attendent d’être découverts et développés en tant que technologies utiles.”
Adaptation naturelle
Les enzymes programmables, en particulier celles qui utilisent un guide ARN, peuvent être rapidement adaptées à différentes utilisations. Par exemple, les enzymes CRISPR utilisent naturellement un guide ARN pour cibler les envahisseurs viraux, mais les biologistes peuvent diriger Cas9 vers n’importe quelle cible en générant leur propre guide ARN. “Il est si facile de simplement modifier une séquence de guidage et de définir une nouvelle cible”, déclare Soumya Kannan, étudiante diplômée du MIT en génie biologique et co-premier auteur de l’article. « Donc, l’une des questions générales qui nous intéresse est d’essayer de voir si d’autres systèmes naturels utilisent ce même type de mécanisme. »
Les premiers indices selon lesquels les protéines OMEGA pourraient être dirigées par l’ARN provenaient des gènes des protéines appelées IscB. Les IscB ne sont pas impliqués dans l’immunité CRISPR et n’étaient pas connus pour s’associer à l’ARN, mais ils ressemblaient à de petites enzymes coupant l’ADN. L’équipe a découvert que chaque IscB avait un petit ARN codé à proximité et il a dirigé les enzymes IscB pour couper des séquences d’ADN spécifiques. Ils ont nommé ces ARN “ωARN”.
Les expériences de l’équipe ont montré que deux autres classes de petites protéines connues sous le nom d’IsrB et de TnpB, l’un des gènes les plus abondants chez les bactéries, utilisent également des ωARN qui servent de guides pour diriger le clivage de l’ADN.

Han Altae-Tran, étudiant diplômé du laboratoire de Zhang, est co-auteur d’un récent article scientifique sur OMEGAS avec Soumya Kannan. Crédit : Avec l’aimable autorisation du laboratoire Zhang
IscB, IsrB et TnpB se trouvent dans des éléments génétiques mobiles appelés transposons. Han Altae-Tran, étudiant diplômé du MIT en génie biologique et co-premier auteur de l’article, explique que chaque fois que ces transposons se déplacent, ils créent un nouvel ARN guide, permettant à l’enzyme qu’ils codent de couper ailleurs.
Il n’est pas clair comment les bactéries bénéficient de ce brassage génomique – ou si elles le font du tout. Les transposons sont souvent considérés comme des morceaux d’ADN égoïstes, préoccupés uniquement par leur propre mobilité et leur propre conservation, dit Kannan. Mais si les hôtes peuvent « coopter » ces systèmes et les réutiliser, les hôtes peuvent acquérir de nouvelles capacités, comme avec les systèmes CRISPR qui confèrent une immunité adaptative.
Les IscB et les TnpB semblent être les prédécesseurs des systèmes CRISPR Cas9 et Cas12. L’équipe soupçonne qu’ils, avec IsrB, ont probablement également donné naissance à d’autres enzymes guidées par l’ARN – et ils sont impatients de les trouver. Ils sont curieux de connaître la gamme de fonctions qui pourraient être exécutées dans la nature par les enzymes guidées par l’ARN, dit Kannan, et l’évolution suspecte probablement déjà profité des enzymes OMEGA comme IscBs et TnpBs pour résoudre les problèmes que les biologistes souhaitent résoudre.
“Beaucoup de choses auxquelles nous avons pensé peuvent déjà exister naturellement dans une certaine mesure”, explique Altae-Tran. “Les versions naturelles de ces types de systèmes pourraient être un bon point de départ pour s’adapter à cette tâche particulière.”
L’équipe s’intéresse également à retracer l’évolution des systèmes guidés par l’ARN plus loin dans le passé. « Trouver tous ces nouveaux systèmes met en lumière l’évolution des systèmes guidés par l’ARN, mais nous ne savons pas d’où vient l’activité guidée par l’ARN elle-même », déclare Altae-Tran. Comprendre ces origines, dit-il, pourrait ouvrir la voie au développement d’encore plus de classes d’outils programmables.
Référence : « La famille de transposons IS200/IS605 très répandue code diverses endonucléases programmables guidées par l’ARN » par Han Altae-Tran, Soumya Kannan, F. Esra Demircioglu, Rachel Oshiro, Suchita P. Nety, Luke J. McKay, Mensur Dlakić, William P Inskeep, Kira S. Makarova, Rhiannon K. Macrae, Eugene V. Koonin et Feng Zhang, 1er octobre 2021, Science.
DOI : 10.1126/science.abj6856
Ce travail a été rendu possible grâce au soutien du Simons Center for the Social Brain du MIT, des National Institutes of Health et de son programme de recherche intra-muros, Howard Hughes Medical Institute, Open Philanthropy, G. Harold et Leila Y. Mathers Charitable Foundation, Edward Mallinckrodt , Jr. Foundation, Poitras Center for Psychiatric Disorders Research au MIT, Hock E. Tan et K. Lisa Yang Center for Autism Research au MIT, Yang-Tan Center for Molecular Therapeutics au MIT, Lisa Yang, famille Phillips, R. Metcalfe, et J. et P. Poitras.